Tratamiento del sarcoma de Ewing y los sarcomas indiferenciados de células redondas pequeñas de hueso y tejido blando (PDQ®)–Versión para profesionales de salud
Información general sobre el sarcoma de Ewing y los sarcomas indiferenciados de células redondas pequeñas de hueso y tejido blando
Se han logrado mejoras notables en la supervivencia de niños y adolescentes con cáncer.[1] Entre 1975 y 2020, la mortalidad por cáncer infantil disminuyó en más de un 50 %.[1-3] La tasa de supervivencia a 5 años del sarcoma de Ewing, aumentó del 59 % a un intervalo del 80 % al 85 % en los niños menores de 15 años y del 20 % al 69 % en los adolescentes de 15 a 19 años.[1,2]
En estudios en los que se usaron marcadores inmunohistoquímicos,[4] análisis citogenéticos,[5,6] análisis genéticos moleculares y cultivos de tejidos [7] se indica que el sarcoma de Ewing se origina a partir de una célula madre mesenquimatosa primordial derivada de la médula ósea.[8,9] Los términos más antiguos, como tumor neuroectodérmico primitivo periférico, tumor de Askin (sarcoma de Ewing de la pared torácica) y sarcoma de Ewing extraóseo (a menudo combinados en el término tumores de la familia del sarcoma de Ewing) se refieren al mismo tumor.
La clasificación de tumores de tejido blando y de hueso de la Organización Mundial de la Salud (OMS) se modificó en 2020 para introducir un capítulo nuevo sobre los sarcomas indiferenciados de células redondas pequeñas de hueso y tejido blando. En este capítulo se incluye el sarcoma de Ewing y tres categorías principales: los sarcomas de células redondas que presentan fusiones de EWSR1 con genes que no pertenecen a la familia ETS, el sarcoma con reordenamiento de CIC y los sarcomas con alteraciones genéticas en BCOR.[10]
Antes de la disponibilidad generalizada de las pruebas genómicas, el sarcoma de Ewing se identificaba mediante análisis microscópico, por la presencia de células redondas pequeñas de color azul, y tinción inmunohistoquímica positiva para CD99. La detección de la translocación t(11;22) recurrente en la mayoría de los tumores de sarcoma de Ewing llevó al descubrimiento de que la mayor parte de los tumores clasificados como sarcoma de Ewing exhibían una translocación de yuxtaposición de una porción del gen EWSR1 y una porción de un gen miembro de la familia ETS, lo que producía un transcrito modificado. No todos los sarcomas indiferenciados de células redondas pequeñas de hueso y tejido blando exhiben esta translocación. Mediante una investigación más profunda, se identificaron otros cambios genéticos, como tumores que exhiben translocaciones que afectan el gen CIC o el gen BCOR. Este grupo de tumores es mucho menos frecuente que el sarcoma de Ewing y la información de estos pacientes se basa en muestras más pequeñas y un tratamiento menos homogéneo; por consiguiente, es difícil medir de manera precisa los desenlaces de los pacientes. La mayoría de los pacientes con estos tumores han recibido tratamiento con regímenes diseñados para el sarcoma de Ewing; y hubo acuerdo en que estos pacientes a menudo se incluyeron en ensayos clínicos de tratamiento del sarcoma de Ewing; en ocasiones, llamado sarcoma de Ewing negativo para translocaciones. En la actualidad, se acepta que estos tumores se diferencian lo suficiente del sarcoma de Ewing y se deben estratificar y analizar de forma independiente de dicho sarcoma, aunque el tratamiento sea similar. En este resumen, estos tumores se describen por separado. Para obtener más información sobre los grupos de tumores menos comunes, consultar las siguientes secciones:
- Sarcomas indiferenciados de células redondas pequeñas con alteraciones genéticas en BCOR.
- Sarcomas indiferenciados de células redondas pequeñas con alteraciones genéticas en CIC.
- Sarcomas indiferenciados de células redondas pequeñas que presentan fusiones de EWSR1 con genes que no pertenecen a la familia ETS.
Incidencia
En los Estados Unidos, entre 2016 y 2020, el National Childhood Cancer Registry (NCCR) notificó una tasa de incidencia de sarcoma de Ewing y sarcomas óseos relacionados de 3,0 casos por millón en niños y adolescentes menores de 20 años.[2] Esta incidencia no modificó la notificada entre 1973 y 2004.[11] Las tasas de incidencia por grupos de edad en la población pediátrica de los Estados Unidos para el sarcoma de Ewing y los sarcomas óseos relacionados se muestran en el Cuadro 1 y la Figura 1. Si bien se han descrito casos bien caracterizados de sarcoma de Ewing en neonatos y lactantes, la incidencia es baja en lactantes y niños pequeños y aumenta en adolescentes.[12,13]
| Edad (años) | Tasa por 1 000 000 | Intervalo de confianza 95 % |
|---|---|---|
| aFuente: National Childhood Cancer Registry (NCCR) Explorer.[2] | ||
| <1 | 0,5 | 0,2–1,1 |
| 1–4 | 1 | 0,7–1,3 |
| 5–9 | 2,3 | 1,9–2,6 |
| 10–14 | 4,3 | 3,9–4,9 |
| 15–19 | 4,5 | 4,0–5,0 |
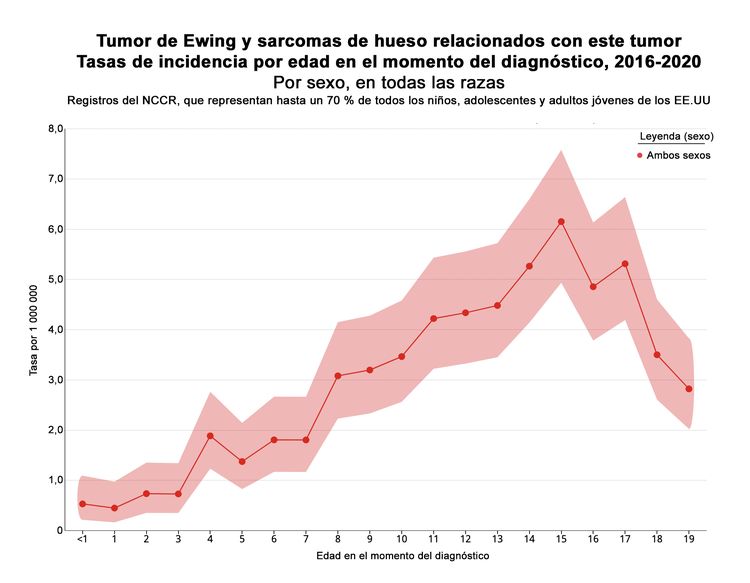
La incidencia del sarcoma de Ewing en los Estados Unidos es 9 veces mayor en las personas blancas que en las personas negras, y la incidencia es intermedia en las personas asiáticas.[14,15] La relativa poca frecuencia del sarcoma de Ewing en las personas de ascendencia africana o asiática se puede explicar, en parte, por un polimorfismo específico en el gen EGR2.[16]
Según los datos obtenidos de 1426 pacientes que participaron en los European Intergroup Cooperative Ewing Sarcoma Studies, el 59 % de los pacientes son varones y el 41 % son mujeres.[17] Estos resultados coinciden con la distribución entre hombres y mujeres del 58 % y el 42 % en los Estados Unidos (edad <20 años) en el conjunto de datos del NCCR (tasa de incidencia de 3,5 y 2,5 casos por millón en hombres y mujeres, respectivamente).[2]
Predisposición genética al sarcoma de Ewing
La comprensión convencional del sarcoma de Ewing causado por una translocación indica que estos pacientes no presentan una predisposición genética[18] Se realizó un análisis retrospectivo controlado por casos de ascendencia global centrado en Europa. El objetivo del estudio fue detectar enriquecimiento de variantes germinales patogénicas en 141 genes conocidos por su predisposición al cáncer entre 1147 pacientes pediátricos con diagnóstico de sarcomas (226 sarcomas de Ewing, 438 osteosarcomas, 180 rabdomiosarcomas y 303 casos de otros sarcomas) y se compararon los resultados con los de personas del grupo de control sin cáncer procesados de forma idéntica. Se encontró una distribución característica de las variantes patogénicas del sarcoma de Ewing, en comparación con otros tipos de sarcoma. El gen FANCC fue el único que exhibió una señal de enriquecimiento para las variantes patogénicas heterocigotas en la cohorte de descubrimiento europea de sarcoma de Ewing (3 personas, oportunidad relativa [OR], 12,6; intervalo de confianza [IC] 95 %, 3,0–43,2; P = 0,003; tasa de descubrimientos falsos, 0,40). Este enriquecimiento de las variantes patogénicas heterocigotas de FANCC también se observó en la cohorte de validación europea de sarcoma de Ewing (3 personas; OR, 7,0; IC 95 %, 1,7–23,6; P = 0,014).
En los estudios de asociación del genoma completo se identificaron locus de susceptibilidad al sarcoma de Ewing en 1p36.22, 10q21 y 15q15.[16,19,20] La secuenciación avanzada a través de la región 10q21.3 identificó un polimorfismo en el gen EGR2, que parece cooperar y magnificar la actividad mejorada del producto génico del gen de fusión EWSR1::FLI1 que se observa en la mayoría de los pacientes con sarcoma de Ewing.[16] El polimorfismo relacionado con el aumento del riesgo se encuentra con una frecuencia mucho más alta en las personas blancas que en las personas negras o asiáticas, lo que posiblemente contribuya a la epidemiología de la infrecuencia relativa del sarcoma de Ewing en estas últimas poblaciones. Se identificaron 3 nuevos locus de susceptibilidad en 6p25.1, 20p11.22 y 20p11.23.[20]
Cuadro clínico inicial
El cuadro clínico inicial del sarcoma de Ewing varía y depende del tamaño y la ubicación del tumor.
Los sitios primarios de enfermedad ósea se enumeran en el Cuadro 2.[21]
| Sitio primario | Tasa de incidencia |
|---|---|
| Cráneo | 5 % |
| Columna vertebral | 7 % |
| Costilla | 11 % |
| Esternón, omóplato y clavícula | 5 % |
| Húmero | 7 % |
| Radio, cúbito, mano | 2 % |
| Pelvis | 18 % |
| Fémur | 11 % |
| Tibia, peroné, rótula, pie | 14 % |
| Tejido blando | 19 % |
El intervalo de tiempo desde el primer síntoma hasta el diagnóstico de sarcoma de Ewing suele ser prolongado, con una mediana notificada de 2 a 5 meses. Los intervalos más prolongados se relacionan con una edad avanzada y sitios primarios pélvicos. El tiempo transcurrido desde el primer síntoma hasta el diagnóstico no se ha relacionado con metástasis, desenlace quirúrgico ni supervivencia.[22]
Cerca del 25 % de los pacientes con sarcoma de Ewing tienen enfermedad metastásica en el momento del diagnóstico; los sitios metastásicos más comunes son los pulmones, los huesos y la médula ósea.[11]
En un análisis retrospectivo se examinaron pacientes tratados en 2 estudios del Children's Oncology Group (COG), INT-0154 y AEWS0031 (NCT00006734). En este estudio se compararon las características clínicas de 213 pacientes con sarcoma de Ewing primario extraesquelético con las de 826 pacientes con sarcoma de Ewing primario óseo.[23] Fue más probable que los pacientes con tumores extraesqueléticos no fueran blancos, presentaran tumores primarios axiales y tuvieran tumores más pequeños que los pacientes con sarcoma de Ewing primario óseo.
La base de datos del Programa de Vigilancia, Epidemiología y Resultados Finales (SEER) se usó para comparar pacientes menores de 40 años con sarcoma de Ewing en sitios primarios esqueléticos (óseos) y extraóseos (extraesqueléticos) (consultar el Cuadro 3).[24] Se encontró que los pacientes con sarcoma de Ewing extraóseo presentaban una probabilidad más alta de tener más edad, ser de sexo femenino, no ser de raza blanca y presentar sitios primarios axiales; así como una probabilidad más baja de tener sitios primarios pélvicos, en comparación con los pacientes con sarcoma de Ewing esquelético (óseo).
| Característica | Sarcoma de Ewing extraóseo | Sarcoma de Ewing esquelético | Valor de P |
|---|---|---|---|
| aAdaptación de Applebaum et al.[24] | |||
| Media de edad (intervalo), años | 20 (0–39) | 16 (0–39) | <0,001 |
| Sexo masculino | 53 % | 63 % | <0,001 |
| Raza blanca | 85 % | 93 % | <0,001 |
| Sitios primarios axiales | 73 % | 54 % | <0,001 |
| Sitios primarios pélvicos | 20 % | 27 % | 0,001 |
Evaluación diagnóstica
Para diagnosticar o estadificar un sarcoma de Ewing se usan las siguientes pruebas y procedimientos:
- Examen físico y antecedentes.
- Imágenes por resonancia magnética (IRM) del sitio del tumor primario.
- Tomografía computarizada (TC) del tórax.
- Tomografía por emisión de positrones (TEP).
- Gammagrafía ósea. Tradicionalmente, la gammagrafía ósea se realizaba de manera rutinaria para la estadificación en todos los pacientes con sarcoma de Ewing. Sin embargo, muchos investigadores creen que la TEP puede reemplazar a la gammagrafía ósea.[25,26]
- Aspiración de médula ósea y biopsia.
- Radiografía de sitios óseos primarios.
- Recuento sanguíneo completo.
- Estudios bioquímicos de la sangre, como la concentración de lactato–deshidrogenasa (LDH).
La evaluación de la metástasis discontinua es importante para los tumores primarios de hueso apendicular. Por lo tanto, las imágenes de todo el hueso comprometido se toman de manera estandarizada. En un estudio retrospectivo, se observó metástasis discontinua en el 15,8 % de los pacientes. La presencia de metástasis discontinua se relacionó con un aumento del riesgo de enfermedad metastásica a distancia.[27]
Se puede considerar la omisión de la biopsia de la médula ósea y la aspiración cuando se usan pruebas con imágenes mediante TEP con flúor F 18-fludesoxiglucosa (18F-FDG) en pacientes que después de los estudios de estadificación iniciales presentan, enfermedad localizada . En una revisión sistemática de estudios de sarcoma de Ewing se evaluó la incidencia de metástasis en la médula ósea y la función de la TEP con 18F-FDG para detectar dichas metástasis.[28] En la revisión se notificó una incidencia combinada de metástasis en la médula ósea del 4,8 % en todos los pacientes con sarcoma de Ewing recién diagnosticado y del 17,5 % en los pacientes con enfermedad metastásica. Solo el 1,2 % de los pacientes presentaron metástasis en la médula ósea como el único sitio metastásico. En comparación con la aspiración y biopsia de la médula ósea, la TEP con 18F-FDG demostró una sensibilidad combinada del 100 %, una especificidad del 96 %, un valor predictivo de un resultado positivo del 75 % y un valor predictivo de un resultado negativo del 100 % para la detección de metástasis en la médula ósea. Para obtener más información sobre la biopsia diagnóstica, consultar la sección Aspectos generales de las opciones de tratamiento del sarcoma de Ewing.
Factores pronósticos
Los dos tipos principales de factores pronósticos para los pacientes con sarcoma de Ewing se agrupan como sigue:
Factores previos al tratamiento
- Metástasis. La presencia o ausencia de enfermedad metastásica es el factor pronóstico unitario más importante para el desenlace. Cualquier tipo de enfermedad metastásica, identificada por imágenes estándar, o estudio morfológico por aspiración de médula ósea o biopsia, constituye un factor de pronóstico adverso. Se detectan metástasis en el momento del diagnóstico en cerca del 25 % de los pacientes.[11]
Los pacientes con enfermedad metastásica confinada al pulmón tienen un pronóstico mejor que los pacientes con sitios metastásicos extrapulmonares.[29-32] El número de lesiones pulmonares no se correlaciona con el desenlace, pero los pacientes con compromiso pulmonar unilateral tienen un pronóstico mejor que los pacientes con compromiso pulmonar bilateral.[33]
Los pacientes con metástasis solo en los huesos evolucionan mejor que los pacientes con metástasis óseas y pulmonares simultáneas.[34,35]
A partir de un análisis de la base de datos SEER, el compromiso de ganglios linfáticos regionales se relaciona con un desenlace general inferior, en comparación con la ausencia de compromiso ganglionar.[36]
- Sitio del tumor. Los pacientes con sarcoma de Ewing en las extremidades distales tienen desenlaces más favorables. Los pacientes con sarcoma de Ewing en las extremidades proximales tienen un pronóstico intermedio, seguido de los pacientes con sitios centrales o pélvicos.[29,31,32,37] Sin embargo, en un ensayo del COG se observaron desenlaces similares en pacientes con tumores primarios pélvicos, en comparación con otros sitios.[21]
En un estudio se analizó de manera retrospectiva la experiencia de una sola institución con el sarcoma de Ewing visceral. Este estudio se enfocó en el tratamiento quirúrgico y en la comparación de desenlaces de pacientes con sarcoma de Ewing visceral y pacientes con sarcoma de Ewing de hueso y tejidos blandos.[38] Se identificaron 156 pacientes con sarcoma de Ewing: 117 casos eran de huesos, 20 de tejidos blandos y 19 de vísceras. Los sarcomas de Ewing viscerales surgieron en los riñones (n = 5), pulmones (n = 5), intestinos (n = 2), esófago (n = 1), hígado (n = 1), páncreas (n = 1), glándulas suprarrenales (n = 1), vagina (n = 1), encéfalo (n = 1) y médula espinal (n = 1). El sarcoma de Ewing visceral fue metastásico con mayor frecuencia en el momento de la presentación (63,2 %; P = 0,005). No obstante, no hubo diferencia significativa en la supervivencia general (SG) o la supervivencia sin recaída entre los grupos de sarcoma de Ewing, con intervalos de seguimiento similares.
- Tumores primarios esqueléticos versus extraesqueléticos. El COG dirigió un análisis retrospectivo de 2 ensayos de cooperación grandes donde se administraron regímenes de tratamiento parecidos.[23] Se identificaron 213 pacientes con tumores primarios extraesqueléticos y 826 pacientes con tumores primarios esqueléticos. Fue más probable que los pacientes con tumores primarios extraesqueléticos presentaran un sitio primario axial y menos probable que tuvieran tumores primarios grandes, en comparación con los pacientes con tumores primarios esqueléticos; además los primeros presentaron un pronóstico significativamente más favorable.
- Tamaño o volumen del tumor. En la mayoría de los estudios se observó que el tamaño o el volumen del tumor son un factor pronóstico importante. Para definir los tumores más grandes se usa un límite de volumen de 100 ml o 200 ml, o una sola dimensión mayor de 8 cm. Los tumores más grandes tienden a aparecer en lugares desfavorables.[31,32,39]
- Edad: Los pacientes más jóvenes suelen tener mejor pronóstico que los de más edad, como se observa en los siguientes estudios:[13,29,32,37,40-42]
- En estudios realizados en América del Norte, los pacientes menores de 10 años tuvieron un mejor desenlace que aquellos de 10 a 17 años en el momento del diagnóstico (riesgo relativo [RR], 1,4). Los pacientes mayores de 18 años presentaron un desenlace inferior (RR, 2,5).[43-45]
- En una revisión retrospectiva de 2 ensayos alemanes consecutivos de pacientes con sarcoma de Ewing, se identificó a 47 pacientes mayores de 40 años.[46] Con una terapia multimodal adecuada, la supervivencia fue comparable a la observada en adolescentes tratados en los mismos ensayos.
- En una revisión de la base de datos SEER, de 1973 a 2011, se identificaron 1957 pacientes con sarcoma de Ewing.[47] De los pacientes, 39 (2 %) tenían menos de 12 meses en el momento del diagnóstico. Fue menos probable que los lactantes recibieran radioterapia y más probable que presentaran sitios primarios en tejidos blandos. La muerte prematura fue más frecuente en los lactantes, pero la SG no difirió significativamente de la supervivencia de los pacientes de más edad.
- En una revisión retrospectiva europea se identificaron 2635 pacientes con sarcoma de Ewing óseo.[48] Los sitios de tumores primarios y metastásicos difirieron según los grupos de edad de niños pequeños (0–9 años), adolescencia temprana (10–14 años), adolescencia tardía (15–19 años), adultos jóvenes (20–24 años) y adultos (mayores de 24 años). Los niños pequeños exhibían las diferencias más notables con respecto al sitio de la enfermedad, con una proporción más baja de tumores primarios pélvicos y tumores axiales. Los niños pequeños también presentaron con menos frecuencia enfermedad metastásica en el momento del diagnóstico.
- Sexo. Las mujeres con sarcoma de Ewing tienen mejor pronóstico que los hombres.[14,32,37]
- LDH sérica. Las concentraciones elevadas de LDH antes del tratamiento se relacionan con un pronóstico inferior. El aumento de las concentraciones de LDH también se relaciona con tumores primarios grandes y enfermedad metastásica.[37]
- Fractura patológica. En un análisis retrospectivo de una sola institución de 78 pacientes con sarcoma de Ewing, se indicó que la fractura patológica en el momento de la presentación inicial se relacionó con una supervivencia sin complicaciones (SSC) y una SG inferiores.[49][Nivel de evidencia C1] En otro estudio se encontró que la fractura patológica en el momento del diagnóstico no impedía la resección quirúrgica y no se relacionaba con un desenlace adverso.[50]
- Tratamiento oncológico previo. En la base de datos SEER, se diagnosticó a 58 pacientes con sarcoma de Ewing después del tratamiento de una neoplasia maligna previa (2,1 % de los pacientes con sarcoma de Ewing). Estos pacientes se compararon con 2756 pacientes con sarcoma de Ewing como primer cáncer durante el mismo período. Los pacientes cuyo sarcoma de Ewing era una neoplasia maligna secundaria tenían más años (sarcoma de Ewing secundario, mediana de edad de 47,8 años; sarcoma de Ewing primario, mediana de edad de 22,5 años), fue más probable que presentaran un tumor primario en un sitio axial o extraesquelético y tuvieron un pronóstico más precario (tasas de SG a 5 años del 43,5 % en los pacientes con sarcoma de Ewing secundario y del 64,2 % en los pacientes con sarcoma de Ewing primario).[51]
-
Alteraciones cromosómicas.
- Un cariotipo complejo (definido como la presencia de 5 o más anomalías cromosómicas independientes en el momento del diagnóstico) y cifras cromosómicas modales menores de 50 son factores de pronóstico adverso.[52]
- La ganancia del cromosoma 1q o la deleción del cromosoma 16q se han relacionado con un pronóstico más precario en los pacientes con sarcoma de Ewing de varias cohortes.[53-55] Estas dos alteraciones cromosómicas a menudo se presentan juntas en varios tipos de cáncer, entre ellos, el sarcoma de Ewing.[56] Es probable que la presencia simultánea se deba a que su causa sea una translocación t(1;16) desequilibrada que produce ganancia del cromosoma 1q junto con pérdida de material cromosómico de 16q.[57,58]
- Células detectables del sarcoma de Ewing, transcritos de fusión o DNA tumoral circulante (ctDNA) en sangre periférica: Se han propuesto varios métodos para evaluar la presencia del sarcoma de Ewing en la sangre periférica. La citometría de flujo para las células que expresan el antígeno CD99 no fue lo suficientemente sensible como para servir como biomarcador confiable.[59,60] Tampoco se consideró a la reacción en cadena de la polimerasa con retrotranscripción (RT-PCR) para la traslocación EWSR1::FLI1 como un biomarcador fiable.[61]
En una técnica más sensible se utilizaron cebadores específicos para el paciente creados después de la identificación del punto de interrupción específico de la translocación en combinación con PCR de gotas digitales para detectar la fusión EWSR1. Mediante el uso de esta técnica se notificó un umbral de sensibilidad del 0,009 % al 0,018 %.[62] Las concentraciones de DNA libre circulante fueron más altas en los pacientes con enfermedad metastásica que en los pacientes con enfermedad localizada.
Se detectó la fusión de EWSR1 en ctDNA mediante un ensayo de secuenciación de captura híbrida de última generación y un ensayo de secuenciación de genoma completo de muy baja frecuencia en plasma conservado de pacientes con sarcoma de Ewing. Entre los pacientes con sarcoma de Ewing localizado recién diagnosticado, el ctDNA detectable se relacionó con tasas inferiores de SSC a 3 años (48,6 vs. 82,1 %; P= 0,006) y tasas de SG (79,8 vs. 92,6 %; P= 0,01).[63]
El ctDNA se evaluó por separado en 102 pacientes tratados en el ensayo EWING2008 (NCT00987636) mediante la PCR de gotas digitales.[64] Los números de copia del ctDNA previos al tratamiento se correlacionaron con la SSC y la SG. Se observó una reducción en la concentración de ctDNA por debajo del límite de detección en la mayoría de los pacientes después de dos bloques de quimioterapia de inducción con vincristina, ifosfamida, doxorrubicina y etopósido (VIDE). La persistencia de ctDNA después de dos bloques de VIDE fue un factor de predicción fuerte de desenlace precario.
- Detección de transcritos de fusión en médula con características morfológicas normales. La RT-PCR se puede usar para detectar transcritos de fusión en la médula ósea. En un solo estudio retrospectivo en pacientes con médula de características morfológicas normales y sin ningún otro sitio metastásico, la detección de un transcrito de fusión en la médula o la sangre periférica se relacionó con un aumento del riesgo de recaída.[60] Sin embargo, en una cohorte más grande (n = 225) de pacientes con sarcoma de Ewing localizado no se observó ninguna diferencia en la SSC o la SG según la detección de transcritos de fusión en sangre o médula ósea.[65]
- Alteraciones génicas. Se realizó un análisis prospectivo de variantes de TP53 o deleciones de CDKN2A en pacientes con sarcoma de Ewing inscritos en ensayos clínicos del COG. En el análisis no se encontró relación de estas alteraciones con la SSC.[66]
En un estudio de 299 pacientes con sarcoma de Ewing, 41 pacientes (14 %) tenían variantes de STAG2 y 16 pacientes (5 %) tenían variantes de TP53.[55] No hubo relación con la SG en los pacientes con variantes de STAG2 o TP53 solas. No obstante, los 9 pacientes (3 %) con tumores que tenían ambas variantes de STAG2 y TP53 exhibieron una disminución significativa en la tasa de SG (<20 % al cabo de 4 años).
El COG analizó la expresión de STAG2 mediante una prueba inmunohistoquímica en niños con sarcoma de Ewing que participaron en ensayos de tratamiento de primera línea.[67] Se observó la perdida de STAG2 en 29 de 108 pacientes con enfermedad localizada y en 6 de 27 pacientes con enfermedad metastásica. En los pacientes que se sometieron a una prueba inmunohistoquímica y secuenciación, ningún caso (0 de 17) con expresión de STAG2 presentó variantes de STAG2 y 2 de 7 casos con pérdida de STAG2 presentaron variantes de STAG2. Entre los pacientes con enfermedad localizada, la tasa de SSC a 5 años fue del 54 % (IC 95 %, 34–70 %) en aquellos con pérdida de STAG2, en comparación con el 75 % (IC 95 %, 63–84 %) en aquellos con expresión de STAG2 (P = 0,0034).
Los siguientes aspectos no se consideran factores de pronóstico adverso del sarcoma de Ewing:
- Características histopatológicas. El grado de diferenciación neural no constituye un factor pronóstico del sarcoma de Ewing.[68,69]
- Subtipo de fusión. Una translocación EWSR1::ETS que se relaciona con el sarcoma de Ewing puede ocurrir en varios sitios de ruptura posibles en cada uno de los genes que se unen para formar el nuevo segmento de DNA. Aunque se pensaba que el sitio era un aspecto importante,[70] en dos series grandes se observó que el sitio del punto de ruptura de la translocación EWSR1::ETS no es un factor de pronóstico precario.[71,72]
Factores de respuesta al tratamiento inicial
En múltiples estudios se observó que los pacientes con tumor viable mínimo o sin tumor residual después de la quimioterapia prequirúrgica tienen una SSC significativamente mejor que los pacientes con cantidades más grandes de tumor viable.[21,73-76]; [77][Nivel de evidencia C2] En particular, los pacientes con enfermedad localizada que no tienen tumor viable en el momento de la cirugía de control local presentan desenlaces muy favorables.[21]; [77][Nivel de evidencia C2] El sexo femenino y la edad más temprana predicen una buena respuesta histológica a la terapia preoperatoria.[78] En los pacientes que se someten a TEP antes y después de la quimioterapia de preinducción y posinducción, la disminución de la absorción en la TEP después de la quimioterapia se correlacionó con una buena respuesta histológica y un mejor desenlace.[79-81]
Los pacientes con respuesta precaria a la quimioterapia antes de la cirugía tienen más riesgo de presentar recidiva local.[82]
Se realizó un análisis retrospectivo de los factores de riesgo para la recidiva en pacientes que recibieron quimioterapia inicial y se sometieron a resección quirúrgica del tumor primario.[83][Nivel de evidencia C1] Al cabo de una mediana de seguimiento de 7,6 años en 982 pacientes se notificaron los siguientes aspectos:
- Los factores de riesgo adverso para una recidiva local fueron tumores primarios pélvicos (cociente de riesgos instantáneos [CRI], 2,04; IC 95 %, 1,10–3,80) y una resección marginal o intralesional (CRI, 2,28; IC 95 %, 1,25–4,16). La adición de radioterapia se relacionó con la mejora del desenlace (CRI, 0,52; IC 95 %, 0,28–0,95).
- Los factores de riesgo adverso para una nueva metástasis pulmonar fueron la presencia de necrosis inferior al 90 % (CRI, 2,13; IC 95 %, 1,13–4,00) y el antecedente de metástasis pulmonar (CRI, 4,90; IC 95 %, 2,28–8,52).
- Los factores de riesgo adverso para muerte fueron metástasis pulmonar (CRI, 8,08; IC 95 %, 4,01–16,29), metástasis ósea o metástasis de otro tipo (CRI, 10,23; IC 95 %, 4,90–21,36) y necrosis inferior al 90 % (CRI, 6,35; IC 95 %, 3,18–12,69).
- La recidiva local temprana (0–24 meses) influyó de manera adversa en la supervivencia (CRI, 3,79; IC 95 %, 1,34–10,76).
En una cohorte retrospectiva de 148 pacientes con sarcoma de Ewing metastásico pulmonar, el 41,2 % presentó resolución radiográfica de los nódulos pulmonares después de la quimioterapia de inducción inicial.[84] Estos pacientes tuvieron una SG superior en comparación con los pacientes que tenían nódulos residuales al final de la inducción (71,2 vs. 50,2 % a los 5 años). Se observaron desenlaces particularmente favorables en los pacientes que presentaron resolución temprana de nódulos pulmonares y recibieron radioterapia de consolidación dirigida a todo el pulmón (tasa de SG a 5 años, 85,2 %).
Bibliografía
- Smith MA, Altekruse SF, Adamson PC, et al.: Declining childhood and adolescent cancer mortality. Cancer 120 (16): 2497-506, 2014. [PUBMED Abstract]
- National Cancer Institute: NCCR*Explorer: An interactive website for NCCR cancer statistics. Bethesda, MD: National Cancer Institute. Available online. Last accessed February 25, 2025.
- Surveillance Research Program, National Cancer Institute: SEER*Explorer: An interactive website for SEER cancer statistics. Bethesda, MD: National Cancer Institute. Available online. Last accessed December 30, 2024.
- Olsen SH, Thomas DG, Lucas DR: Cluster analysis of immunohistochemical profiles in synovial sarcoma, malignant peripheral nerve sheath tumor, and Ewing sarcoma. Mod Pathol 19 (5): 659-68, 2006. [PUBMED Abstract]
- Delattre O, Zucman J, Melot T, et al.: The Ewing family of tumors--a subgroup of small-round-cell tumors defined by specific chimeric transcripts. N Engl J Med 331 (5): 294-9, 1994. [PUBMED Abstract]
- Dagher R, Pham TA, Sorbara L, et al.: Molecular confirmation of Ewing sarcoma. J Pediatr Hematol Oncol 23 (4): 221-4, 2001. [PUBMED Abstract]
- Llombart-Bosch A, Carda C, Peydro-Olaya A, et al.: Soft tissue Ewing's sarcoma. Characterization in established cultures and xenografts with evidence of a neuroectodermic phenotype. Cancer 66 (12): 2589-601, 1990. [PUBMED Abstract]
- Suvà ML, Riggi N, Stehle JC, et al.: Identification of cancer stem cells in Ewing's sarcoma. Cancer Res 69 (5): 1776-81, 2009. [PUBMED Abstract]
- Tirode F, Laud-Duval K, Prieur A, et al.: Mesenchymal stem cell features of Ewing tumors. Cancer Cell 11 (5): 421-9, 2007. [PUBMED Abstract]
- Choi JH, Ro JY: The 2020 WHO Classification of Tumors of Soft Tissue: Selected Changes and New Entities. Adv Anat Pathol 28 (1): 44-58, 2021. [PUBMED Abstract]
- Esiashvili N, Goodman M, Marcus RB: Changes in incidence and survival of Ewing sarcoma patients over the past 3 decades: Surveillance Epidemiology and End Results data. J Pediatr Hematol Oncol 30 (6): 425-30, 2008. [PUBMED Abstract]
- Kim SY, Tsokos M, Helman LJ: Dilemmas associated with congenital ewing sarcoma family tumors. J Pediatr Hematol Oncol 30 (1): 4-7, 2008. [PUBMED Abstract]
- van den Berg H, Dirksen U, Ranft A, et al.: Ewing tumors in infants. Pediatr Blood Cancer 50 (4): 761-4, 2008. [PUBMED Abstract]
- Jawad MU, Cheung MC, Min ES, et al.: Ewing sarcoma demonstrates racial disparities in incidence-related and sex-related differences in outcome: an analysis of 1631 cases from the SEER database, 1973-2005. Cancer 115 (15): 3526-36, 2009. [PUBMED Abstract]
- Beck R, Monument MJ, Watkins WS, et al.: EWS/FLI-responsive GGAA microsatellites exhibit polymorphic differences between European and African populations. Cancer Genet 205 (6): 304-12, 2012. [PUBMED Abstract]
- Grünewald TG, Bernard V, Gilardi-Hebenstreit P, et al.: Chimeric EWSR1-FLI1 regulates the Ewing sarcoma susceptibility gene EGR2 via a GGAA microsatellite. Nat Genet 47 (9): 1073-8, 2015. [PUBMED Abstract]
- Paulussen M, Craft AW, Lewis I, et al.: Results of the EICESS-92 Study: two randomized trials of Ewing's sarcoma treatment--cyclophosphamide compared with ifosfamide in standard-risk patients and assessment of benefit of etoposide added to standard treatment in high-risk patients. J Clin Oncol 26 (27): 4385-93, 2008. [PUBMED Abstract]
- Gillani R, Camp SY, Han S, et al.: Germline predisposition to pediatric Ewing sarcoma is characterized by inherited pathogenic variants in DNA damage repair genes. Am J Hum Genet 109 (6): 1026-1037, 2022. [PUBMED Abstract]
- Postel-Vinay S, Véron AS, Tirode F, et al.: Common variants near TARDBP and EGR2 are associated with susceptibility to Ewing sarcoma. Nat Genet 44 (3): 323-7, 2012. [PUBMED Abstract]
- Machiela MJ, Grünewald TGP, Surdez D, et al.: Genome-wide association study identifies multiple new loci associated with Ewing sarcoma susceptibility. Nat Commun 9 (1): 3184, 2018. [PUBMED Abstract]
- Leavey PJ, Laack NN, Krailo MD, et al.: Phase III Trial Adding Vincristine-Topotecan-Cyclophosphamide to the Initial Treatment of Patients With Nonmetastatic Ewing Sarcoma: A Children's Oncology Group Report. J Clin Oncol 39 (36): 4029-4038, 2021. [PUBMED Abstract]
- Brasme JF, Chalumeau M, Oberlin O, et al.: Time to diagnosis of Ewing tumors in children and adolescents is not associated with metastasis or survival: a prospective multicenter study of 436 patients. J Clin Oncol 32 (18): 1935-40, 2014. [PUBMED Abstract]
- Cash T, McIlvaine E, Krailo MD, et al.: Comparison of clinical features and outcomes in patients with extraskeletal versus skeletal localized Ewing sarcoma: A report from the Children's Oncology Group. Pediatr Blood Cancer 63 (10): 1771-9, 2016. [PUBMED Abstract]
- Applebaum MA, Worch J, Matthay KK, et al.: Clinical features and outcomes in patients with extraskeletal Ewing sarcoma. Cancer 117 (13): 3027-32, 2011. [PUBMED Abstract]
- Tal AL, Doshi H, Parkar F, et al.: The Utility of 18FDG PET/CT Versus Bone Scan for Identification of Bone Metastases in a Pediatric Sarcoma Population and a Review of the Literature. J Pediatr Hematol Oncol 43 (2): 52-58, 2021. [PUBMED Abstract]
- Costelloe CM, Chuang HH, Daw NC: PET/CT of Osteosarcoma and Ewing Sarcoma. Semin Roentgenol 52 (4): 255-268, 2017. [PUBMED Abstract]
- Saifuddin A, Michelagnoli M, Pressney I: Skip metastases in appendicular Ewing sarcoma: relationship to distant metastases at diagnosis, chemotherapy response and overall survival. Skeletal Radiol 52 (3): 585-591, 2023. [PUBMED Abstract]
- Campbell KM, Shulman DS, Grier HE, et al.: Role of bone marrow biopsy for staging new patients with Ewing sarcoma: A systematic review. Pediatr Blood Cancer 68 (2): e28807, 2021. [PUBMED Abstract]
- Cotterill SJ, Ahrens S, Paulussen M, et al.: Prognostic factors in Ewing's tumor of bone: analysis of 975 patients from the European Intergroup Cooperative Ewing's Sarcoma Study Group. J Clin Oncol 18 (17): 3108-14, 2000. [PUBMED Abstract]
- Miser JS, Krailo MD, Tarbell NJ, et al.: Treatment of metastatic Ewing's sarcoma or primitive neuroectodermal tumor of bone: evaluation of combination ifosfamide and etoposide--a Children's Cancer Group and Pediatric Oncology Group study. J Clin Oncol 22 (14): 2873-6, 2004. [PUBMED Abstract]
- Rodríguez-Galindo C, Liu T, Krasin MJ, et al.: Analysis of prognostic factors in ewing sarcoma family of tumors: review of St. Jude Children's Research Hospital studies. Cancer 110 (2): 375-84, 2007. [PUBMED Abstract]
- Karski EE, McIlvaine E, Segal MR, et al.: Identification of Discrete Prognostic Groups in Ewing Sarcoma. Pediatr Blood Cancer 63 (1): 47-53, 2016. [PUBMED Abstract]
- Paulussen M, Ahrens S, Craft AW, et al.: Ewing's tumors with primary lung metastases: survival analysis of 114 (European Intergroup) Cooperative Ewing's Sarcoma Studies patients. J Clin Oncol 16 (9): 3044-52, 1998. [PUBMED Abstract]
- Paulussen M, Ahrens S, Burdach S, et al.: Primary metastatic (stage IV) Ewing tumor: survival analysis of 171 patients from the EICESS studies. European Intergroup Cooperative Ewing Sarcoma Studies. Ann Oncol 9 (3): 275-81, 1998. [PUBMED Abstract]
- Ladenstein R, Pötschger U, Le Deley MC, et al.: Primary disseminated multifocal Ewing sarcoma: results of the Euro-EWING 99 trial. J Clin Oncol 28 (20): 3284-91, 2010. [PUBMED Abstract]
- Applebaum MA, Goldsby R, Neuhaus J, et al.: Clinical features and outcomes in patients with Ewing sarcoma and regional lymph node involvement. Pediatr Blood Cancer 59 (4): 617-20, 2012. [PUBMED Abstract]
- Bacci G, Longhi A, Ferrari S, et al.: Prognostic factors in non-metastatic Ewing's sarcoma tumor of bone: an analysis of 579 patients treated at a single institution with adjuvant or neoadjuvant chemotherapy between 1972 and 1998. Acta Oncol 45 (4): 469-75, 2006. [PUBMED Abstract]
- Wallace MW, Niec JA, Ghani MOA, et al.: Distribution and Surgical Management of Visceral Ewing Sarcoma Among Children and Adolescents. J Pediatr Surg 58 (9): 1727-1735, 2023. [PUBMED Abstract]
- Ahrens S, Hoffmann C, Jabar S, et al.: Evaluation of prognostic factors in a tumor volume-adapted treatment strategy for localized Ewing sarcoma of bone: the CESS 86 experience. Cooperative Ewing Sarcoma Study. Med Pediatr Oncol 32 (3): 186-95, 1999. [PUBMED Abstract]
- De Ioris MA, Prete A, Cozza R, et al.: Ewing sarcoma of the bone in children under 6 years of age. PLoS One 8 (1): e53223, 2013. [PUBMED Abstract]
- Huh WW, Daw NC, Herzog CE, et al.: Ewing sarcoma family of tumors in children younger than 10 years of age. Pediatr Blood Cancer 64 (4): , 2017. [PUBMED Abstract]
- Ahmed SK, Randall RL, DuBois SG, et al.: Identification of Patients With Localized Ewing Sarcoma at Higher Risk for Local Failure: A Report From the Children's Oncology Group. Int J Radiat Oncol Biol Phys 99 (5): 1286-1294, 2017. [PUBMED Abstract]
- Grier HE, Krailo MD, Tarbell NJ, et al.: Addition of ifosfamide and etoposide to standard chemotherapy for Ewing's sarcoma and primitive neuroectodermal tumor of bone. N Engl J Med 348 (8): 694-701, 2003. [PUBMED Abstract]
- Granowetter L, Womer R, Devidas M, et al.: Dose-intensified compared with standard chemotherapy for nonmetastatic Ewing sarcoma family of tumors: a Children's Oncology Group Study. J Clin Oncol 27 (15): 2536-41, 2009. [PUBMED Abstract]
- Womer RB, West DC, Krailo MD, et al.: Randomized controlled trial of interval-compressed chemotherapy for the treatment of localized Ewing sarcoma: a report from the Children's Oncology Group. J Clin Oncol 30 (33): 4148-54, 2012. [PUBMED Abstract]
- Pieper S, Ranft A, Braun-Munzinger G, et al.: Ewing's tumors over the age of 40: a retrospective analysis of 47 patients treated according to the International Clinical Trials EICESS 92 and EURO-E.W.I.N.G. 99. Onkologie 31 (12): 657-63, 2008. [PUBMED Abstract]
- Wong T, Goldsby RE, Wustrack R, et al.: Clinical features and outcomes of infants with Ewing sarcoma under 12 months of age. Pediatr Blood Cancer 62 (11): 1947-51, 2015. [PUBMED Abstract]
- Worch J, Ranft A, DuBois SG, et al.: Age dependency of primary tumor sites and metastases in patients with Ewing sarcoma. Pediatr Blood Cancer 65 (9): e27251, 2018. [PUBMED Abstract]
- Schlegel M, Zeumer M, Prodinger PM, et al.: Impact of Pathological Fractures on the Prognosis of Primary Malignant Bone Sarcoma in Children and Adults: A Single-Center Retrospective Study of 205 Patients. Oncology 94 (6): 354-362, 2018. [PUBMED Abstract]
- Bramer JA, Abudu AA, Grimer RJ, et al.: Do pathological fractures influence survival and local recurrence rate in bony sarcomas? Eur J Cancer 43 (13): 1944-51, 2007. [PUBMED Abstract]
- Applebaum MA, Goldsby R, Neuhaus J, et al.: Clinical features and outcomes in patients with secondary Ewing sarcoma. Pediatr Blood Cancer 60 (4): 611-5, 2013. [PUBMED Abstract]
- Roberts P, Burchill SA, Brownhill S, et al.: Ploidy and karyotype complexity are powerful prognostic indicators in the Ewing's sarcoma family of tumors: a study by the United Kingdom Cancer Cytogenetics and the Children's Cancer and Leukaemia Group. Genes Chromosomes Cancer 47 (3): 207-20, 2008. [PUBMED Abstract]
- Hattinger CM, Pötschger U, Tarkkanen M, et al.: Prognostic impact of chromosomal aberrations in Ewing tumours. Br J Cancer 86 (11): 1763-9, 2002. [PUBMED Abstract]
- Mackintosh C, Ordóñez JL, García-Domínguez DJ, et al.: 1q gain and CDT2 overexpression underlie an aggressive and highly proliferative form of Ewing sarcoma. Oncogene 31 (10): 1287-98, 2012. [PUBMED Abstract]
- Tirode F, Surdez D, Ma X, et al.: Genomic landscape of Ewing sarcoma defines an aggressive subtype with co-association of STAG2 and TP53 mutations. Cancer Discov 4 (11): 1342-53, 2014. [PUBMED Abstract]
- Mrózek K, Bloomfield CD: Der(16)t(1;16) is a secondary chromosome aberration in at least eighteen different types of human cancer. Genes Chromosomes Cancer 23 (1): 78-80, 1998. [PUBMED Abstract]
- Mugneret F, Lizard S, Aurias A, et al.: Chromosomes in Ewing's sarcoma. II. Nonrandom additional changes, trisomy 8 and der(16)t(1;16). Cancer Genet Cytogenet 32 (2): 239-45, 1988. [PUBMED Abstract]
- Hattinger CM, Rumpler S, Ambros IM, et al.: Demonstration of the translocation der(16)t(1;16)(q12;q11.2) in interphase nuclei of Ewing tumors. Genes Chromosomes Cancer 17 (3): 141-50, 1996. [PUBMED Abstract]
- Dubois SG, Epling CL, Teague J, et al.: Flow cytometric detection of Ewing sarcoma cells in peripheral blood and bone marrow. Pediatr Blood Cancer 54 (1): 13-8, 2010. [PUBMED Abstract]
- Schleiermacher G, Peter M, Oberlin O, et al.: Increased risk of systemic relapses associated with bone marrow micrometastasis and circulating tumor cells in localized ewing tumor. J Clin Oncol 21 (1): 85-91, 2003. [PUBMED Abstract]
- Zoubek A, Ladenstein R, Windhager R, et al.: Predictive potential of testing for bone marrow involvement in Ewing tumor patients by RT-PCR: a preliminary evaluation. Int J Cancer 79 (1): 56-60, 1998. [PUBMED Abstract]
- Shukla NN, Patel JA, Magnan H, et al.: Plasma DNA-based molecular diagnosis, prognostication, and monitoring of patients with EWSR1 fusion-positive sarcomas. JCO Precis Oncol 2017: , 2017. [PUBMED Abstract]
- Shulman DS, Klega K, Imamovic-Tuco A, et al.: Detection of circulating tumour DNA is associated with inferior outcomes in Ewing sarcoma and osteosarcoma: a report from the Children's Oncology Group. Br J Cancer 119 (5): 615-621, 2018. [PUBMED Abstract]
- Krumbholz M, Eiblwieser J, Ranft A, et al.: Quantification of Translocation-Specific ctDNA Provides an Integrating Parameter for Early Assessment of Treatment Response and Risk Stratification in Ewing Sarcoma. Clin Cancer Res 27 (21): 5922-5930, 2021. [PUBMED Abstract]
- Vo KT, Edwards JV, Epling CL, et al.: Impact of Two Measures of Micrometastatic Disease on Clinical Outcomes in Patients with Newly Diagnosed Ewing Sarcoma: A Report from the Children's Oncology Group. Clin Cancer Res 22 (14): 3643-50, 2016. [PUBMED Abstract]
- Lerman DM, Monument MJ, McIlvaine E, et al.: Tumoral TP53 and/or CDKN2A alterations are not reliable prognostic biomarkers in patients with localized Ewing sarcoma: a report from the Children's Oncology Group. Pediatr Blood Cancer 62 (5): 759-65, 2015. [PUBMED Abstract]
- Shulman DS, Chen S, Hall D, et al.: Adverse prognostic impact of the loss of STAG2 protein expression in patients with newly diagnosed localised Ewing sarcoma: A report from the Children's Oncology Group. Br J Cancer 127 (12): 2220-2226, 2022. [PUBMED Abstract]
- Parham DM, Hijazi Y, Steinberg SM, et al.: Neuroectodermal differentiation in Ewing's sarcoma family of tumors does not predict tumor behavior. Hum Pathol 30 (8): 911-8, 1999. [PUBMED Abstract]
- Luksch R, Sampietro G, Collini P, et al.: Prognostic value of clinicopathologic characteristics including neuroectodermal differentiation in osseous Ewing's sarcoma family of tumors in children. Tumori 85 (2): 101-7, 1999 Mar-Apr. [PUBMED Abstract]
- de Alava E, Kawai A, Healey JH, et al.: EWS-FLI1 fusion transcript structure is an independent determinant of prognosis in Ewing's sarcoma. J Clin Oncol 16 (4): 1248-55, 1998. [PUBMED Abstract]
- van Doorninck JA, Ji L, Schaub B, et al.: Current treatment protocols have eliminated the prognostic advantage of type 1 fusions in Ewing sarcoma: a report from the Children's Oncology Group. J Clin Oncol 28 (12): 1989-94, 2010. [PUBMED Abstract]
- Le Deley MC, Delattre O, Schaefer KL, et al.: Impact of EWS-ETS fusion type on disease progression in Ewing's sarcoma/peripheral primitive neuroectodermal tumor: prospective results from the cooperative Euro-E.W.I.N.G. 99 trial. J Clin Oncol 28 (12): 1982-8, 2010. [PUBMED Abstract]
- Paulussen M, Ahrens S, Dunst J, et al.: Localized Ewing tumor of bone: final results of the cooperative Ewing's Sarcoma Study CESS 86. J Clin Oncol 19 (6): 1818-29, 2001. [PUBMED Abstract]
- Rosito P, Mancini AF, Rondelli R, et al.: Italian Cooperative Study for the treatment of children and young adults with localized Ewing sarcoma of bone: a preliminary report of 6 years of experience. Cancer 86 (3): 421-8, 1999. [PUBMED Abstract]
- Wunder JS, Paulian G, Huvos AG, et al.: The histological response to chemotherapy as a predictor of the oncological outcome of operative treatment of Ewing sarcoma. J Bone Joint Surg Am 80 (7): 1020-33, 1998. [PUBMED Abstract]
- Oberlin O, Deley MC, Bui BN, et al.: Prognostic factors in localized Ewing's tumours and peripheral neuroectodermal tumours: the third study of the French Society of Paediatric Oncology (EW88 study). Br J Cancer 85 (11): 1646-54, 2001. [PUBMED Abstract]
- Lozano-Calderón SA, Albergo JI, Groot OQ, et al.: Complete tumor necrosis after neoadjuvant chemotherapy defines good responders in patients with Ewing sarcoma. Cancer 129 (1): 60-70, 2023. [PUBMED Abstract]
- Ferrari S, Bertoni F, Palmerini E, et al.: Predictive factors of histologic response to primary chemotherapy in patients with Ewing sarcoma. J Pediatr Hematol Oncol 29 (6): 364-8, 2007. [PUBMED Abstract]
- Hawkins DS, Schuetze SM, Butrynski JE, et al.: [18F]Fluorodeoxyglucose positron emission tomography predicts outcome for Ewing sarcoma family of tumors. J Clin Oncol 23 (34): 8828-34, 2005. [PUBMED Abstract]
- Denecke T, Hundsdörfer P, Misch D, et al.: Assessment of histological response of paediatric bone sarcomas using FDG PET in comparison to morphological volume measurement and standardized MRI parameters. Eur J Nucl Med Mol Imaging 37 (10): 1842-53, 2010. [PUBMED Abstract]
- Palmerini E, Colangeli M, Nanni C, et al.: The role of FDG PET/CT in patients treated with neoadjuvant chemotherapy for localized bone sarcomas. Eur J Nucl Med Mol Imaging 44 (2): 215-223, 2017. [PUBMED Abstract]
- Lin PP, Jaffe N, Herzog CE, et al.: Chemotherapy response is an important predictor of local recurrence in Ewing sarcoma. Cancer 109 (3): 603-11, 2007. [PUBMED Abstract]
- Bosma SE, Rueten-Budde AJ, Lancia C, et al.: Individual risk evaluation for local recurrence and distant metastasis in Ewing sarcoma: A multistate model: A multistate model for Ewing sarcoma. Pediatr Blood Cancer 66 (11): e27943, 2019. [PUBMED Abstract]
- Reiter AJ, Huang L, Craig BT, et al.: Survival outcomes in pediatric patients with metastatic Ewing sarcoma who achieve a rapid complete response of pulmonary metastases. Pediatr Blood Cancer 71 (7): e31026, 2024. [PUBMED Abstract]
Clasificación celular del sarcoma de Ewing
El sarcoma de Ewing pertenece al grupo de neoplasias que se conocen, por lo general, como tumores de células pequeñas, redondas y azules de la niñez. Las células del sarcoma de Ewing contienen núcleos redondos u ovalados con cromatina fina y dispersa sin nucléolos. En ocasiones, hay células con núcleos más pequeños, más hipercromáticos y probablemente degenerativos, que producen una configuración de células claras y oscuras. El citoplasma varía en cantidad. En los casos clásicos, el citoplasma es trasparente y contiene glucógeno, que se puede destacar con una tinción de reacción del ácido peryódico de Schiff. Las células tumorales están muy apretujadas y crecen en un patrón difuso sin signos de organización estructural. Los tumores con la translocación necesaria para que presenten diferenciación neuronal no se consideran como una entidad separada, sino parte de un proceso continuo de diferenciación.
La proteína de membrana CD99 se expresa en la mayoría de los casos de sarcoma de Ewing y resulta útil para diagnosticar estos tumores cuando los resultados se interpretan en el contexto de parámetros clínicos y patológicos.[1] La positividad para CD99 no es exclusiva del sarcoma de Ewing; dicha positividad determinada mediante inmunoquímica se encuentra en varios tumores, como el sarcoma sinovial, el linfoma no Hodgkin y los tumores del estroma gastrointestinal. NKX2.2 es un antígeno nuclear que también se evalúa con frecuencia en la prueba inmunohistoquímica para respaldar el diagnóstico de sarcoma de Ewing, aunque tampoco es 100 % específico para este diagnóstico.[2]
Para obtener más información sobre la clasificación celular de otros sarcomas indiferenciados de células redondas pequeñas, consultar la sección Sarcomas indiferenciados de células redondas pequeñas (similares al Ewing).
Bibliografía
- Parham DM, Hijazi Y, Steinberg SM, et al.: Neuroectodermal differentiation in Ewing's sarcoma family of tumors does not predict tumor behavior. Hum Pathol 30 (8): 911-8, 1999. [PUBMED Abstract]
- Yoshida A, Sekine S, Tsuta K, et al.: NKX2.2 is a useful immunohistochemical marker for Ewing sarcoma. Am J Surg Pathol 36 (7): 993-9, 2012. [PUBMED Abstract]
Características genómicas del sarcoma de Ewing
Características moleculares del sarcoma de Ewing
La Organización Mundial de la Salud identificó la presencia de una fusión génica que involucra EWSR1 o FUS y un gen de la familia ETS como un elemento que define el sarcoma de Ewing.[1] El gen EWSR1, ubicado en la banda q12 del cromosoma 22, es un miembro de la familia FET (FUS, EWSR1, TAF15) de proteínas de unión al RNA.[2] De manera característica, el extremo amino del gen EWSR1 se yuxtapone con el extremo carboxílico de un gen de la familia ETS de factores de transcripción de unión al DNA (ver el Cuadro 4). El gen FLI1, ubicado en la banda q24 del cromosoma 11, es miembro de la familia ETS y es el compañero de fusión de la familia ETS para EWSR1 en el 85 % al 90 % de los casos pediátricos de sarcoma de Ewing.[3-5] Otros miembros de la familia ETS que se combinan con el gen EWSR1 son ERG, ETV1, ETV4 y FEV.[6] En raras ocasiones, FUS, otro miembro de la familia FET, puede sustituir a EWSR1.[7] Por último, hay algunos casos poco frecuentes en los que la translocación de EWSR1 se produce con genes que no son miembros de la familia de oncogenes ETS. Se cree que estos tumores son distintos del sarcoma de Ewing y se analizan por separado. Para obtener más información, consultar la sección Sarcomas indiferenciados de células redondas pequeñas (similares al Ewing).
Una translocación EWSR1::FLI1 que se relaciona con el sarcoma de Ewing puede ocurrir en varios sitios de ruptura posibles en cada uno de los genes que se unen para formar el nuevo segmento de DNA. A pesar de que en algún momento se pensó que era importante,[8] en dos series grandes se observó que el sitio de ruptura de la translocación EWSR1::FLI1 no es un factor de pronóstico adverso.[9,10]
Además de las anomalías sistemáticas que comprometen el gen EWSR1, en la mayoría de los casos de sarcoma de Ewing se observan anomalías numéricas y estructurales secundarias. Las ganancias cromosómicas son más comunes que las pérdidas cromosómicas, y también se observan desequilibrios cromosómicos estructurales.[11] Dos de las anomalías cromosómicas más comunes son las que afectan el cromosoma 8 o los cromosomas 1 y 16.[11]
- Ganancia de un cromosoma 8 completo (trisomía 8). La trisomía 8 es la alteración cromosómica más frecuente en el sarcoma de Ewing y se presenta en casi el 50 % de los tumores.[3,4] La ganancia del cromosoma 8 no presenta importancia pronóstica.[3,12]
- Ganancia del cromosoma 1q y pérdida del cromosoma 16q. Estas anomalías ocurren en aproximadamente el 20 % de los pacientes y, a menudo, se presentan juntas. La ganancia del cromosoma 1q o la deleción del cromosoma 16q se ha relacionado con un pronóstico más precario en los pacientes con sarcoma de Ewing de varias cohortes.[3,13,14] Estas dos alteraciones cromosómicas a menudo se presentan juntas en varios tipos de cáncer, entre ellos, el sarcoma de Ewing.[15] Es probable que la presencia simultánea se deba a que su causa es una translocación t(1;16) desequilibrada que produce ganancia del cromosoma 1q junto con pérdida de material cromosómico de 16q.[12,16]
El panorama genómico del sarcoma de Ewing se caracteriza por un genoma relativamente inactivo, con una escasez de variantes en las vías que podrían ser susceptibles de tratamiento con terapias dirigidas nuevas.[3-5] Las alteraciones genómicas recurrentes se describen a continuación. Para algunas de estas alteraciones genómicas, se han hecho afirmaciones de importancia pronóstica. Sin embargo, estas afirmaciones se deben considerar con cautela debido al tamaño pequeño de la mayoría de los estudios, la baja frecuencia de muchas de las alteraciones genómicas, el uso variable del tejido tumoral de las muestras del momento del diagnóstico versus las de la recaída, y la necesidad de considerar factores clínicos de pronóstico, como el tamaño del tumor y la presencia de enfermedad metastásica.
- Variantes de STAG2. Las variantes de STAG2, un miembro del complejo de la cohesina, se presentan en cerca del 15 % al 20 % de los casos.[3-5] Estas variantes conducen a la pérdida de la expresión y el funcionamiento de STAG2 en las células tumorales.[5] Se observó pérdida de la expresión de STAG2 (detectada mediante pruebas inmunohistoquímicas [IHQ]) en tumores en los que no se puede detectar una variante de STAG2. En un informe, la pérdida de expresión de STAG2, comprobada mediante pruebas IHQ, se relacionó con un pronóstico inferior.[17]
- Deleciones de CDKN2A. Se observaron deleciones de CDKN2A en un 12 % a un 22 % de los casos.[3-5]
- Variantes de TP53. Se identificaron variantes de TP53 en cerca de un 6 % a un 7 % de los casos de sarcoma de Ewing notificados por equipos de investigación en pediatría.[3-5] Se han descrito tasas más elevadas de variantes de TP53 (hasta en un 19 %) en cohortes de una sola institución que contienen porcentajes más altos de pacientes adultos.[18,19] En un informe retrospectivo, la coexistencia de variantes de STAG2 y TP53 se relacionó con un desenlace clínico precario.
- Alteraciones de ERF. Se notificaron alteraciones genómicas de ERF que condujeron a la pérdida de función (mutación en el marco de lectura, mutación de cambio de sentido y deleción importante) en el 7 % de los tumores de sarcoma de Ewing.[18] En un segundo informe, se observaron alteraciones de ERF a una tasa del 3 % en otra cohorte de sarcoma de Ewing.[4]
- Otros genes con alteraciones genómicas recurrentes en el sarcoma de Ewing. Se notificaron alteraciones genómicas recurrentes en menos del 5 % de los pacientes con sarcoma de Ewing: EZH2,[3,19] BCOR,[3] SMARCA4,[19] CREBBP,[19] TERT y FGFR1.[18]
Todas las traslocaciones del sarcoma de Ewing se pueden encontrar mediante análisis citogenético estándar. En la actualidad, con frecuencia se realiza un análisis rápido de hibridación fluorescente in situ (FISH) en busca de una ruptura del gen EWSR1 para confirmar el diagnóstico molecular de sarcoma de Ewing.[20] Sin embargo, el resultado de esta prueba se debe considerar con cautela. En los sarcomas de Ewing que albergan las traslocaciones FUS se obtendrán resultados negativos en las pruebas porque no hay translocación del gen EWSR1. Además, otros tumores de células pequeñas y redondas también contienen translocaciones de diferentes miembros de la familia ETS con EWSR1, como el tumor desmoplásico de células pequeñas redondas, el sarcoma de células claras, el condrosarcoma mixoide extraesquelético y el liposarcoma mixoide, que a veces dan resultados positivos cuando se someten a FISH con sonda de escisión para EWSR1. En un análisis minucioso de 85 pacientes con tumores de células pequeñas redondas y azules sin reordenamiento de EWSR1 se identificaron 8 pacientes con reordenamiento de FUS mediante el uso de FISH con una sonda de escisión para EWSR1.[21] De los pacientes con fusiones EWSR1::ERG, 4 no se detectaron mediante FISH con una sonda de escisión para EWSR1. Los autores recomiendan no confiar de forma exclusiva en las sondas de escisión para EWSR1 durante el análisis de tumores de células pequeñas redondas y azules con gran positividad inmunohistoquímica frente al CD99. Los ensayos de secuenciación de última generación, incluso los paneles de fusión específicos, se utilizan ahora con frecuencia para la evaluación de estos tumores.
| Miembro de la familia FET | Fusión con un oncogén recíproco similar a ETS | Translocación | Comentario |
|---|---|---|---|
| aEstos oncogenes recíprocos no son miembros de la familia de oncogenes ETS; por lo tanto, estos tumores no se clasifican como sarcoma de Ewing. | |||
| EWSR1 | EWSR1::FLI1 | t(11;22)(q24;q12) | Más común; alrededor del 85 % al 90 % de los casos |
| EWSR1::ERG | t(21;22)(q22;q12) | Más común en segundo término; alrededor del 10 % de los casos | |
| EWSR1::ETV1 | t(7;22)(p22;q12) | Poco frecuente | |
| EWSR1::ETV4 | t(17;22)(q12;q12) | Poco frecuente | |
| EWSR1::FEV | t(2;22)(q35;q12) | Poco frecuente | |
| EWSR1::NFATC2a | t(20;22)(q13;q12) | Poco frecuente | |
| EWSR1::POU5F1a | t(6;22)(p21;q12) | ||
| EWSR1::SMARCA5a | t(4;22)(q31;q12) | Poco frecuente | |
| EWSR1::PATZ1a | t(6;22)(p21;q12) | ||
| EWSR1::SP3a | t(2;22)(q31;q12) | Poco frecuente | |
| FUS | FUS::ERG | t(16;21)(p11;q22) | Poco frecuente |
| FUS::FEV | t(2;16)(q35;p11) | Poco frecuente | |
Bibliografía
- WHO Classification of Tumours Editorial Board: WHO Classification of Tumours. Volume 3: Soft Tissue and Bone Tumours. 5th ed., IARC Press, 2020.
- Schwartz JC, Cech TR, Parker RR: Biochemical Properties and Biological Functions of FET Proteins. Annu Rev Biochem 84: 355-79, 2015. [PUBMED Abstract]
- Tirode F, Surdez D, Ma X, et al.: Genomic landscape of Ewing sarcoma defines an aggressive subtype with co-association of STAG2 and TP53 mutations. Cancer Discov 4 (11): 1342-53, 2014. [PUBMED Abstract]
- Crompton BD, Stewart C, Taylor-Weiner A, et al.: The genomic landscape of pediatric Ewing sarcoma. Cancer Discov 4 (11): 1326-41, 2014. [PUBMED Abstract]
- Brohl AS, Solomon DA, Chang W, et al.: The genomic landscape of the Ewing Sarcoma family of tumors reveals recurrent STAG2 mutation. PLoS Genet 10 (7): e1004475, 2014. [PUBMED Abstract]
- Hattinger CM, Rumpler S, Strehl S, et al.: Prognostic impact of deletions at 1p36 and numerical aberrations in Ewing tumors. Genes Chromosomes Cancer 24 (3): 243-54, 1999. [PUBMED Abstract]
- Sankar S, Lessnick SL: Promiscuous partnerships in Ewing's sarcoma. Cancer Genet 204 (7): 351-65, 2011. [PUBMED Abstract]
- de Alava E, Kawai A, Healey JH, et al.: EWS-FLI1 fusion transcript structure is an independent determinant of prognosis in Ewing's sarcoma. J Clin Oncol 16 (4): 1248-55, 1998. [PUBMED Abstract]
- van Doorninck JA, Ji L, Schaub B, et al.: Current treatment protocols have eliminated the prognostic advantage of type 1 fusions in Ewing sarcoma: a report from the Children's Oncology Group. J Clin Oncol 28 (12): 1989-94, 2010. [PUBMED Abstract]
- Le Deley MC, Delattre O, Schaefer KL, et al.: Impact of EWS-ETS fusion type on disease progression in Ewing's sarcoma/peripheral primitive neuroectodermal tumor: prospective results from the cooperative Euro-E.W.I.N.G. 99 trial. J Clin Oncol 28 (12): 1982-8, 2010. [PUBMED Abstract]
- Roberts P, Burchill SA, Brownhill S, et al.: Ploidy and karyotype complexity are powerful prognostic indicators in the Ewing's sarcoma family of tumors: a study by the United Kingdom Cancer Cytogenetics and the Children's Cancer and Leukaemia Group. Genes Chromosomes Cancer 47 (3): 207-20, 2008. [PUBMED Abstract]
- Hattinger CM, Rumpler S, Ambros IM, et al.: Demonstration of the translocation der(16)t(1;16)(q12;q11.2) in interphase nuclei of Ewing tumors. Genes Chromosomes Cancer 17 (3): 141-50, 1996. [PUBMED Abstract]
- Hattinger CM, Pötschger U, Tarkkanen M, et al.: Prognostic impact of chromosomal aberrations in Ewing tumours. Br J Cancer 86 (11): 1763-9, 2002. [PUBMED Abstract]
- Mackintosh C, Ordóñez JL, García-Domínguez DJ, et al.: 1q gain and CDT2 overexpression underlie an aggressive and highly proliferative form of Ewing sarcoma. Oncogene 31 (10): 1287-98, 2012. [PUBMED Abstract]
- Mrózek K, Bloomfield CD: Der(16)t(1;16) is a secondary chromosome aberration in at least eighteen different types of human cancer. Genes Chromosomes Cancer 23 (1): 78-80, 1998. [PUBMED Abstract]
- Mugneret F, Lizard S, Aurias A, et al.: Chromosomes in Ewing's sarcoma. II. Nonrandom additional changes, trisomy 8 and der(16)t(1;16). Cancer Genet Cytogenet 32 (2): 239-45, 1988. [PUBMED Abstract]
- Shulman DS, Chen S, Hall D, et al.: Adverse prognostic impact of the loss of STAG2 protein expression in patients with newly diagnosed localised Ewing sarcoma: A report from the Children's Oncology Group. Br J Cancer 127 (12): 2220-2226, 2022. [PUBMED Abstract]
- Ogura K, Elkrief A, Bowman AS, et al.: Prospective Clinical Genomic Profiling of Ewing Sarcoma: ERF and FGFR1 Mutations as Recurrent Secondary Alterations of Potential Biologic and Therapeutic Relevance. JCO Precis Oncol 6: e2200048, 2022. [PUBMED Abstract]
- Rock A, Uche A, Yoon J, et al.: Bioinformatic Analysis of Recurrent Genomic Alterations and Corresponding Pathway Alterations in Ewing Sarcoma. J Pers Med 13 (10): , 2023. [PUBMED Abstract]
- Monforte-Muñoz H, Lopez-Terrada D, Affendie H, et al.: Documentation of EWS gene rearrangements by fluorescence in-situ hybridization (FISH) in frozen sections of Ewing's sarcoma-peripheral primitive neuroectodermal tumor. Am J Surg Pathol 23 (3): 309-15, 1999. [PUBMED Abstract]
- Chen S, Deniz K, Sung YS, et al.: Ewing sarcoma with ERG gene rearrangements: A molecular study focusing on the prevalence of FUS-ERG and common pitfalls in detecting EWSR1-ERG fusions by FISH. Genes Chromosomes Cancer 55 (4): 340-9, 2016. [PUBMED Abstract]
Información sobre los estadios del sarcoma de Ewing
Los estudios de estadificación que se obtienen antes del tratamiento incluyen los siguientes:
- Imágenes por resonancia magnética (IRM) del sitio primario.
- Tomografías computarizadas (TC) del sitio primario y el tórax.
- Tomografías por emisión de positrones con flúor F 18-fludesoxiglucosa (TEP con 18F-FDG) o TEP-TC con 18F-FDG.
- Tradicionalmente, la gammagrafía ósea forma parte de la evaluación de estadificación del sarcoma de Ewing. Sin embargo, muchos investigadores creen que la TEP puede reemplazar a la gammagrafía ósea.[1,2]
- Aspiración de médula ósea y biopsia.
En pacientes con diagnóstico confirmado de sarcoma de Ewing, los estudios de estadificación previos al tratamiento incluyen una IRM o una TC según el sitio primario. A pesar de que tanto la TC como la IRM son equivalentes en términos de estadificación, el uso de ambos tipos de imágenes puede ayudar a planificar la radioterapia.[3] Es posible que la IRM de todo el cuerpo proporcione información adicional que podría alterar la planificación del tratamiento.[4] Otros estudios de estadificación previa al tratamiento incluyen gammagrafía ósea y TC del tórax. En algunos estudios, la determinación del volumen tumoral previo al tratamiento es una variable importante.
Las TEP-TC con 18F-FDG han demostrado una sensibilidad y especificidad altas en el sarcoma de Ewing y ahora se usan de manera rutinaria para completar la estadificación. En un estudio institucional, la TEP con 18F-FDG exhibió una correlación muy alta con la gammagrafía ósea; los investigadores indicaron que podría reemplazar la gammagrafía ósea durante la evaluación inicial de la enfermedad.[5] Este hallazgo se confirmó en una revisión retrospectiva de una sola institución.[6] La TEP-TC con 18F-FDG es más precisa que la TEP con 18F-FDG sola en el sarcoma de Ewing.[7-9]
La aspiración de la médula ósea y biopsia se ha considerado el estándar de atención del sarcoma de Ewing. No obstante, en dos estudios retrospectivos se observó que en los pacientes (N = 141) evaluados mediante gammagrafía ósea o TEP y TC pulmonar sin evidencia de metástasis, los aspirados de médula ósea y las biopsias fueron negativos en todos los casos.[5,10] En una revisión retrospectiva de una sola institución de 504 pacientes con sarcoma de Ewing se identificaron 12 pacientes con metástasis en la médula ósea.[11] Se encontró que solo 1 paciente tenía compromiso de la médula ósea sin ningún otro sitio de enfermedad metastásica, con una incidencia de 1 por 367 (0,3 %) en pacientes con enfermedad clínicamente localizada. Ahora se cuestiona el uso rutinario de aspiraciones y biopsias de médula ósea en pacientes sin metástasis óseas.
En el caso del sarcoma de Ewing, los tumores se estadifican como localizados o metastásicos, y no se usan con frecuencia otros sistemas de estadificación. En el caso del sarcoma de Ewing, el tumor se define como localizado cuando en la exploración clínica y por imágenes no se encuentra diseminación fuera del sitio primario ni compromiso ganglionar regional. Es posible que se presente extensión continua al tejido blando adyacente. Cuando no está claro si hay compromiso ganglionar regional, se indica una confirmación patológica.
Bibliografía
- Costelloe CM, Chuang HH, Daw NC: PET/CT of Osteosarcoma and Ewing Sarcoma. Semin Roentgenol 52 (4): 255-268, 2017. [PUBMED Abstract]
- Tal AL, Doshi H, Parkar F, et al.: The Utility of 18FDG PET/CT Versus Bone Scan for Identification of Bone Metastases in a Pediatric Sarcoma Population and a Review of the Literature. J Pediatr Hematol Oncol 43 (2): 52-58, 2021. [PUBMED Abstract]
- Meyer JS, Nadel HR, Marina N, et al.: Imaging guidelines for children with Ewing sarcoma and osteosarcoma: a report from the Children's Oncology Group Bone Tumor Committee. Pediatr Blood Cancer 51 (2): 163-70, 2008. [PUBMED Abstract]
- Mentzel HJ, Kentouche K, Sauner D, et al.: Comparison of whole-body STIR-MRI and 99mTc-methylene-diphosphonate scintigraphy in children with suspected multifocal bone lesions. Eur Radiol 14 (12): 2297-302, 2004. [PUBMED Abstract]
- Newman EN, Jones RL, Hawkins DS: An evaluation of [F-18]-fluorodeoxy-D-glucose positron emission tomography, bone scan, and bone marrow aspiration/biopsy as staging investigations in Ewing sarcoma. Pediatr Blood Cancer 60 (7): 1113-7, 2013. [PUBMED Abstract]
- Ulaner GA, Magnan H, Healey JH, et al.: Is methylene diphosphonate bone scan necessary for initial staging of Ewing sarcoma if 18F-FDG PET/CT is performed? AJR Am J Roentgenol 202 (4): 859-67, 2014. [PUBMED Abstract]
- Völker T, Denecke T, Steffen I, et al.: Positron emission tomography for staging of pediatric sarcoma patients: results of a prospective multicenter trial. J Clin Oncol 25 (34): 5435-41, 2007. [PUBMED Abstract]
- Gerth HU, Juergens KU, Dirksen U, et al.: Significant benefit of multimodal imaging: PET/CT compared with PET alone in staging and follow-up of patients with Ewing tumors. J Nucl Med 48 (12): 1932-9, 2007. [PUBMED Abstract]
- Treglia G, Salsano M, Stefanelli A, et al.: Diagnostic accuracy of ¹⁸F-FDG-PET and PET/CT in patients with Ewing sarcoma family tumours: a systematic review and a meta-analysis. Skeletal Radiol 41 (3): 249-56, 2012. [PUBMED Abstract]
- Kopp LM, Hu C, Rozo B, et al.: Utility of bone marrow aspiration and biopsy in initial staging of Ewing sarcoma. Pediatr Blood Cancer 62 (1): 12-5, 2015. [PUBMED Abstract]
- Cesari M, Righi A, Colangeli M, et al.: Bone marrow biopsy in the initial staging of Ewing sarcoma: Experience from a single institution. Pediatr Blood Cancer 66 (6): e27653, 2019. [PUBMED Abstract]
Aspectos generales de las opciones de tratamiento del sarcoma de Ewing
Es importante que los pacientes sean evaluados por especialistas en las disciplinas adecuadas (por ejemplo, oncología médica, cirugía oncológica, ortopedia oncológica y radioncología) tan pronto como sea posible. La revisión multidisciplinaria con radiólogos y patólogos a menudo se realiza en centros especializados en sarcoma.
Antes de obtener la biopsia se deben tomar las imágenes indicadas del sitio primario sospechoso. Se consultará al cirujano (oncólogo u ortopedista) que llevará a cabo la intervención definitiva sobre la ubicación de la incisión para la biopsia con el fin de asegurar que la incisión se realice en un lugar adecuado. La ubicación de la incisión es especialmente importante si se anticipa que la lesión podría extirparse por completo después de un tratamiento sistémico inicial o si se quiere intentar un procedimiento para conservar el miembro. Casi nunca está indicado intentar una resección primaria del sarcoma de Ewing conocido en el diagnóstico inicial. Salvo pocas excepciones, el sarcoma de Ewing es sensible a la quimioterapia y responderá al tratamiento sistémico inicial, lo que reduce el riesgo de diseminación del tumor a los tejidos circundantes y hace que la cirugía final sea más sencilla e inocua. Siempre que sea posible, la biopsia se debe obtener del tejido blando para evitar aumentar el riesgo de fractura.[1] Si la muestra de biopsia inicial se extrae del hueso, es necesario reservar tejido sin descalcificación porque esta desnaturaliza el DNA y resulta imposible obtener el perfil genómico del tejido tumoral.[2] Antes de la biopsia o cirugía, se consulta al patólogo para asegurarse de que la incisión no comprometa el campo de radiación y de que se obtengan múltiples tipos de muestras de tejido adecuadas. Toda vez que sea posible, es importante obtener tejido fresco para los estudios citogenéticos y de patología molecular. Una segunda opción es realizar una biopsia con aguja, siempre que se obtenga tejido adecuado para los estudios moleculares.[3]
En el Cuadro 5 se describen las opciones de tratamiento del sarcoma de Ewing localizado, metastásico y recidivante.
| Grupo de tratamiento | Opciones de tratamiento estándar | |
|---|---|---|
| Sarcoma de Ewing localizado | Quimioterapia | |
| Medidas de control local: | ||
| Cirugía | ||
| Radioterapia | ||
| Dosis altas de quimioterapia con rescate autógeno de células madre | ||
| Sarcoma de Ewing metastásico | Quimioterapia | |
| Cirugía | ||
| Radioterapia | ||
| Sarcoma de Ewing recidivante | Quimioterapia (no se considera un tratamiento estándar) | |
| Cirugía (no se considera un tratamiento estándar) | ||
| Radioterapia (no se considera un tratamiento estándar) | ||
| Quimioterapia de dosis alta con apoyo de células madre (no se considera un tratamiento estándar) | ||
| Otras terapias (no se consideran tratamientos estándar) | ||
El éxito del tratamiento de los pacientes con sarcoma de Ewing requiere quimioterapia sistémica [4-10] en combinación con cirugía o radioterapia para lograr el control local del tumor.[11-15] En general, los pacientes reciben quimioterapia antes de las medidas de control local. En los pacientes que se someten a cirugía, se evalúan los márgenes quirúrgicos y la respuesta histológica para planificar el tratamiento posoperatorio. Los pacientes con enfermedad metastásica suelen tener una buena respuesta inicial a la quimioterapia preoperatoria, pero en la mayoría de los casos, la enfermedad sólo se controla de forma parcial o reaparece.[16-21] Los pacientes que solo presentan metástasis pulmonares tienen mejor pronóstico que aquellos con metástasis óseas o en la médula ósea. El control local adecuado de los sitios metastásicos; en especial, de las metástasis óseas quizás sea una consideración importante.[22]
Quimioterapia para el sarcoma de Ewing
La quimioterapia multifarmacológica para el sarcoma de Ewing siempre incluye vincristina, doxorrubicina, ifosfamida y etopósido. En la mayoría de los protocolos, también se usa ciclofosfamida y en algunos se incorpora dactinomicina. La forma de administración y la intensidad de la dosis de los cursos de ciclofosfamida difiere bastante en diversos protocolos. En un ensayo del European Intergroup Cooperative Ewing Sarcoma Study (EICESS) se indicó que 1,2 g de ciclofosfamida produjeron una supervivencia sin complicaciones (SSC) similar a la de 6 g de ifosfamida en pacientes con enfermedad de riesgo más bajo. En el ensayo también se identificó una tendencia hacia una mejor SSC en los pacientes con sarcoma de Ewing localizado y enfermedad de riesgo más alto cuando el tratamiento incluyó etopósido (GER-GPOH-EICESS-92 [NCT00002516]).[23][Nivel de evidencia A1]
Por lo general, los protocolos en los Estados Unidos alternan cursos de vincristina, ciclofosfamida y doxorrubicina (VDC) con cursos de ifosfamida y etopósido (IE),[8] mediante la reducción de los intervalos.[24-26] Mientras, en los protocolos europeos durante muchos años se utilizó vincristina, doxorrubicina y un alquilante, con etopósido o sin este, como ciclo de tratamiento único.[10] Después de completar el ensayo aleatorizado EURO EWING 2012 (EE2012) (ver más abajo), los investigadores europeos cambiaron a terapia con ciclos de VDC alternados con ciclos de IE.[27][Nivel de evidencia B1] La duración de la quimioterapia primaria oscila entre 6 meses y alrededor de 1 año.
Evidencia (quimioterapia):
- Un consorcio internacional de países europeos condujo el ensayo EURO-EWING-INTERGROUP-EE99 (NCT00020566) entre 2000 y 2010.[28][Nivel de evidencia A1] Todos los pacientes recibieron terapia de inducción con 6 ciclos de vincristina, ifosfamida, doxorrubicina y etopósido (VIDE), seguidos de control local y, luego, un ciclo de vincristina, dactinomicina e ifosfamida (VAI). Los pacientes que se clasificaron como de riesgo estándar si presentaban enfermedad localizada y buena respuesta histológica a la terapia, o que tenían tumores localizados de menos de 200 ml de volumen en el momento de la presentación, se trataron con radioterapia sola como tratamiento local. Los pacientes de riesgo estándar (n = 856) se asignaron al azar a recibir terapia de mantenimiento con 7 ciclos de vincristina, dactinomicina y ciclofosfamida (VAC) o VAI.
- No hubo ninguna diferencia significativa en la SSC o la supervivencia general (SG) entre los pacientes que recibieron VAC y los que recibieron VAI.
- La tasa de SSC a 3 años para esta población de riesgo bajo fue del 77 %.
- Es difícil comparar este desenlace con el de otras series grandes porque la población del estudio excluyó a los pacientes con respuesta precaria a la terapia inicial o aquellos con tumores de más de 200 ml de volumen que recibieron la terapia de control local con radioterapia sola. En todas las otras series publicadas se notifican resultados para todos los pacientes sin metástasis clínicamente detectables en el cuadro clínico inicial; por lo tanto, en estas otras series se incluyeron pacientes con respuesta precaria y tumores primarios más grandes tratados con radioterapia sola que se excluyeron del estudio EURO-EWING-INTERGROUP-EE99.
- En un estudio del Children's Oncology Group (COG) (COG-AEWS0031 [NCT00006734]), los pacientes sin metástasis en el cuadro clínico inicial se asignaron al azar para recibir ciclos de VDC en alternancia con ciclos de IE en intervalos de 2 o 3 semanas.[24]
- La administración de ciclos de VDC/IE a intervalos de 2 semanas produjo una tasa de SSC superior (tasa de SSC a 5 años, 73 %) que la administración de ciclos en alternancia a intervalos de 3 semanas (tasa de SSC a 5 años, 65 %). Tras un seguimiento más prolongado, se confirmó la ventaja de la quimioterapia de intervalos reducidos.[25]
- La tasa de SSC a 10 años fue del 70 % con la quimioterapia de intervalos reducidos, en comparación con el 61 % con la quimioterapia de intervalos estándar (P = 0,03). La tasa de SG a 10 años fue del 76 % con la quimioterapia de intervalos reducidos, en comparación con el 69 % con la quimioterapia de intervalos estándar (P = 0,04).
- El EE2012 fue un ensayo multicéntrico internacional de fase III en el que se incluyeron 2 grupos de tratamiento aleatorizados: el régimen de inducción VIDE europeo y el régimen VDC/IE, que es el estándar de inducción en América del Norte. Los pacientes con sarcoma de Ewing localizado y metastásico eran aptos para el estudio.[27][Nivel de evidencia B1]
- Los cocientes de riesgos instantáneos (CRI) para la SSC (0,71) y la SG (0,62) favorecieron el régimen VDC/IE, en comparación con el VIDE. Las probabilidades posteriores fueron del 99 % para la SSC y la SG, lo cual demostró la superioridad del régimen de VDC/IE.
- Las tasas de neutropenia febril fueron elevadas con el régimen VIDE. No hubo otras diferencias importantes en cuanto a los efectos tóxicos graves entre ambos regímenes.
- La superioridad de VDC/IE sobre VIDE se observó en todos los subgrupos, definidos según la edad del paciente, el sexo, el estadio, el volumen tumoral o el país de residencia.
- El Brazilian Cooperative Study Group llevó a cabo un ensayo multiinstitucional en el que se incorporó carboplatino a un régimen intensivo adaptado al riesgo administrado a 175 niños con sarcoma de Ewing localizado o metastásico.[29][Nivel de evidencia B4]
- Los investigadores encontraron que la adición de carboplatino produjo un aumento significativo de la toxicidad sin una mejora del desenlace.
- El COG dirigió un ensayo prospectivo aleatorizado en pacientes con sarcoma de Ewing localizado. Todos los pacientes recibieron ciclos de VDC y ciclos de IE. Los pacientes se asignaron al azar a recibir, o no, ciclos experimentales adicionales de vincristina, ciclofosfamida y topotecán.[26]
- La tasa de SSC a 5 años fue del 78 % (intervalo de confianza [CI] 95 %, 72–82 %) en los pacientes que recibieron el tratamiento experimental y del 79 % (IC 95 %, 74–83 %) en los pacientes que recibieron el tratamiento estándar.
- El tratamiento experimental no redujo de manera significativa el riesgo de eventos (CRI SSC para el grupo experimental vs. grupo estándar, 0,86; P unilateral = 0,19).
Control local (cirugía y radioterapia) del sarcoma de Ewing
Los abordajes de tratamiento para el sarcoma de Ewing y la intensidad del tratamiento deben ajustarse con el fin de aumentar al máximo el control local y al mismo tiempo reducir al mínimo la morbilidad.
La cirugía es la forma de control local más utilizada.[30] La radioterapia es otra modalidad eficaz para lograr el control local en casos en que las complicaciones funcionales o cosméticas de la cirugía se consideran demasiado altas, según el criterio de cirujanos oncólogos experimentados. Sin embargo, en un esqueleto inmaduro, la radioterapia a veces causa deformidades posteriores que pueden ser más mórbidas que las causadas por una cirugía. Cuando no se anticipa una resección quirúrgica completa con márgenes patológicamente negativos, por lo general no se realiza cirugía y, en su lugar, se usa radiación definitiva. Cuando se encuentran márgenes patológicamente positivos, se indica radioterapia posoperatoria. Es necesario un análisis multidisciplinario entre un radioncólogo experimentado y un cirujano con el fin de determinar las mejores opciones de tratamiento para el control local de cada caso especifico. Para algunas lesiones con resecabilidad limítrofe, se puede utilizar un abordaje combinado de radioterapia preoperatoria seguida de resección.
El momento en que se aplica el control local quizás afecte el desenlace. En una revisión retrospectiva de la National Cancer Database se identificaron 1318 pacientes con sarcoma de Ewing.[31] Los pacientes que iniciaron la terapia local entre las semanas 6 y 15 presentaron una tasa de SG a 5 años del 78,7 % y una tasa de SG a 10 años del 70,3 %, y los pacientes que iniciaron la terapia local después de la semana 16 presentaron una tasa de SG a 5 años del 70,4 % y una tasa de SG a 10 años del 57,1 % (P < 001). La diferencia en la SG según el tiempo transcurrido hasta la terapia local fue más importante en los pacientes que recibieron radioterapia sola.
En los pacientes con sarcoma de Ewing metastásico, el beneficio de la combinación de cirugía y radioterapia en comparación con cualquiera de las terapias solas para el control local es relativamente menos importante porque el pronóstico general de estos pacientes es mucho más precario que el de los pacientes con enfermedad localizada.
No hay ensayos aleatorizados en los que se compare de manera directa la cirugía con la radioterapia y su función relativa sigue siendo polémica. Aunque en series retrospectivas institucionales se indica mejores resultados en términos de control local y supervivencia con el uso de cirugía que con el uso de radioterapia, la mayoría de estos estudios se ven comprometidos por un sesgo de selección. En un análisis en el que se utilizó un puntaje de predisposición para ajustar por las características clínicas que pueden influir en la preferencia por cirugía sola, radiación sola o cirugía combinada con radiación, se demostró que se logra una SSC similar con cada modalidad de tratamiento local luego del ajuste por predisposición.[30] Los datos de pacientes con sarcoma de Ewing primario en la pelvis en un ensayo intergrupal norteamericano no mostraron diferencias en el control local o la supervivencia según la modalidad de control (cirugía sola, radioterapia sola o radiación con cirugía).[32]
En el ensayo EURO-EWING-INTERGROUP-EE99 (NCT00020566) se trató de manera prospectiva a 180 pacientes con tumores primarios pélvicos sin enfermedad metastásica clínicamente detectable.[33][Nivel de evidencia B4] En un análisis retrospectivo de los desenlaces de estos pacientes, se observó una mejora de la supervivencia de los pacientes cuyos tumores se trataron con la combinación de radioterapia y cirugía. En el estudio, los criterios para seleccionar las modalidades de control local no estaban definidos de manera prospectiva y los investigadores no tuvieron acceso a información que les permitiera saber el motivo de las decisiones de tratamiento. En los tumores fuera del sacro, el tratamiento local combinado se relacionó con una probabilidad menor de recidiva (14 % [intervalo de confianza (IC) 95 %, 5–23 %] vs. 33 % [IC 95 %, 19–47 %] a 5 años; P = 0,015) y una probabilidad mayor de SG (72 % [IC 95 %, 61–83 %] vs. 47 % [IC 95 %, 33–62 %] a los 5 años; P = 0,024), en comparación con la cirugía sola. Incluso en un subgrupo de pacientes con márgenes quirúrgicos amplios y buena respuesta histológica al tratamiento de inducción, el tratamiento local combinado se relacionó con una probabilidad más alta de SG (87 % [IC 95 %, 74–100 %] vs. 51 % [IC 95 %, 33–69 %] a 5 años; P = 0,009), en comparación con la cirugía sola. Entre los pacientes con tumores óseos que se sometieron a tratamiento quirúrgico —después de controlar por el sitio del tumor en la pelvis, el volumen tumoral y el estado del margen quirúrgico— aquellos que no se sometieron a una resección total del hueso afectado (CRI, 5,04; IC 95 %, 2,07–12,24; P < 0,001), los que presentaron una respuesta histológica deficiente a la quimioterapia de inducción (CRI, 3,72; IC 95 %, 1,51–9,21; P = 0,004) y los que no recibieron radioterapia adicional (CRI, 4,34; IC 95 %, 1,71–11,05; P = 0,002) tuvieron un riesgo de muerte más alto.
Para los pacientes que se someten a resección macroscópica total con enfermedad residual microscópica, se recomienda una dosis de radioterapia de 50,4 Gy. En los pacientes tratados con radioterapia primaria, la dosis de radiación es de 55,8 Gy (45 Gy dirigidos al volumen tumoral inicial y 10,8 Gy adicionales al volumen posterior a la quimioterapia).[14,34]
Evidencia (radioterapia posoperatoria):
- Los investigadores del St. Jude Children’s Research Hospital informaron sobre 39 pacientes con sarcoma de Ewing localizado que recibieron cirugía y radiación.[14]
- La tasa de fracaso local en los pacientes con márgenes positivos fue del 17 % y la tasa de SG del 71 %.
- La tasa de fracaso local en los pacientes con márgenes negativos fue del 5 % y la tasa de SG fue del 94 %.
- En un estudio italiano retrospectivo grande, 45 Gy de radioterapia adyuvante para pacientes con márgenes inadecuados no mejoraron el control local ni la supervivencia sin enfermedad (SSE).[15]
- Estos investigadores concluyeron que se debe considerar el uso de radioterapia definitiva para los pacientes en los que se anticipa que la cirugía será subóptima.
- En el estudio EURO-EWING-INTERGROUP-EE99 (NCT00020566) se notificaron los desenlaces de 599 pacientes que presentaron enfermedad localizada en el cuadro clínico inicial y que se sometieron a resección quirúrgica después de la quimioterapia inicial con, por lo menos, un 90 % de necrosis del tumor primario.[34][Nivel de evidencia C2] En el protocolo se recomendó la radioterapia posoperatoria para los pacientes con márgenes quirúrgicos inadecuados, tumores primarios vertebrales o tumores torácicos con derrame pleural, pero la decisión de usar la radioterapia posoperatoria la tomó el investigador de cada institución.
- Los pacientes que recibieron radioterapia posoperatoria (n = 142) presentaron menor riesgo de fracaso que los pacientes que no la recibieron, incluso después del control por factores pronóstico conocidos, como edad, sexo, sitio del tumor, respuesta clínica, calidad de la resección y necrosis histológica. La mejora más pronunciada se observó en la disminución del riesgo de recidiva local. La mejora fue superior en los pacientes con tumores grandes (>200 ml) y quienes se consideró que tenían necrosis del 100 % en comparación con los pacientes que tenían una necrosis del 90 % al 100 %.
- Hay una interacción nítida entre el tratamiento sistémico y las modalidades de control local para el control local y la SSE. El régimen de inducción empleado en el estudio EURO-EWING-INTERGROUP-EE99 es menos intenso que el régimen de inducción que se usa en los protocolos contemporáneos del COG, por lo tanto, no es apropiado extrapolar los resultados del estudio EURO-EWING-INTERGROUP-EE99 a otros regímenes de quimioterapia sistémica.
Tumores torácicos primarios
Evidencia (cirugía):
- En los ensayos CWS-81, CWS-86, CWS-91, CWS-96 y CWS-2002P del Cooperative Weichteilsarkom Studiengruppe se notificaron los desenlaces y el respectivo tratamiento de 62 pacientes con sarcoma de Ewing torácico.[35]
- La tasa de SG a 5 años fue del 58,7 % (IC 95 %, 52,7–64,7 %) y la tasa de SSC fue del 52,8 % (IC 95 %, 46,8–58,8 %).
- Los pacientes con tumores intratorácicos (n = 24) tuvieron desenlaces más precarios (tasa de SSC, 37,5 % [IC 95 %, 27,5–37,5 %]) que aquellos con tumores en la pared torácica (n = 38; tasa de SSC, 62,3 % [IC 95 %, 54,3–70,3 %]; P = 0,008).
- Los pacientes de 10 años y menos (n = 38) presentaron mejor supervivencia (tasa de SSC, 65,7 % [IC 95 %, 57,7–73,7 %]) que los pacientes mayores de 10 años (tasa de SSC, 31,3 % [IC 95 %, 21,3–41,3 %]; P = 0,01).
- Un tamaño de tumor de 5 cm o menos (n = 15) se relacionó de modo significativo con una mejor supervivencia (tasa de SSC, 93,3 % [IC 95 %, 87,3–99,3 %]), en comparación con un tumor de más de 5 cm (n = 47; tasa de SSC, 40 % [IC 95 %, 33–47 %]; P = 0,002).
- En 36 pacientes se hicieron resecciones primarias, y el 75 % fueron incompletas, lo que se tradujo en una SSC inferior (P = 0,006).
- En 22 de 40 pacientes se hicieron resecciones secundarias completas.
- El COG revisó el desenlace de 98 pacientes con tumores de pared torácica que se trataron en los ensayos INT-0091 e INT-0154 desde 1988 hasta 1998 y encontró lo siguiente:[36]
- La tasa de SSC a 5 años fue del 56 %.
- Los márgenes negativos fueron más frecuentes en los pacientes que recibieron quimioterapia inicial seguida de resección (41 de 53 pacientes, 77 %) que en los pacientes que se sometieron a cirugía inicial (10 de 20 pacientes, 50 %).
- La administración de radioterapia fue más frecuente en los pacientes que se sometieron a cirugía inicial (71 %) que en aquellos que comenzaron el tratamiento con quimioterapia (48 %).
En resumen, en los pacientes aptos se escoge la cirugía como tratamiento local definitivo, pero se considera que es apropiado administrar radioterapia en pacientes con enfermedad irresecable o aquellos con complicaciones funcionales o cosméticas de la cirugía definitiva. Es necesario evaluar la posibilidad de deterioro funcional o cosmético y compararla con la posibilidad de segundos tumores en el campo de radiación. Se debe considerar la radioterapia adyuvante para los pacientes con enfermedad residual microscópica o márgenes inadecuados.
Cuando la evaluación preoperatoria indica una probabilidad alta de que los márgenes quirúrgicos serán estrechos o positivos para compromiso tumoral, la radioterapia preoperatoria logra achicar el tumor y permite una resección quirúrgica con márgenes limpios.[37]
En múltiples análisis se evaluaron los hallazgos diagnósticos, el tratamiento y los desenlaces de pacientes con lesiones óseas primarias en los siguientes sitios:
Quimioterapia de dosis altas con apoyo de células madre para el sarcoma de Ewing
Para los pacientes con un riesgo alto de recaída con tratamientos convencionales, algunos investigadores usaron quimioterapia de dosis altas con trasplante de células madre hematopoyéticas (TCMH) como tratamiento de consolidación, en un esfuerzo por mejorar el desenlace.[19,55-67]
Evidencia (terapia de dosis altas con apoyo de células madre):
- En un estudio prospectivo, los pacientes con metástasis óseas o de médula ósea en el momento del diagnóstico recibieron quimioterapia intensiva, cirugía o radioterapia y quienes presentaban una buena respuesta inicial se sometieron a TCMH.[60]
- El estudio no mostró beneficio del TCMH cuando se comparó con controles históricos.
- En una revisión retrospectiva para la que se usaron registros internacionales de trasplante de médula ósea, se compararon los desenlaces después del tratamiento, con acondicionamiento de intensidad reducida y con acondicionamiento de intensidad alta seguido de TCMH alogénico, en pacientes con sarcoma de Ewing con riesgo alto de recaída.[68][Nivel de evidencia C1]
- No hubo diferencia en los desenlaces y los autores concluyeron que esto indicaba la ausencia de un efecto clínico importante de injerto contra tumor en las células tumorales del sarcoma de Ewing al usar los abordajes vigentes.
- La función de la terapia de dosis altas con busulfano y melfalán (BuMel) seguida de rescate de células madre se investigó en el ensayo prospectivo aleatorizado EURO-EWING-INTERGROUP-EE99 (NCT00020566) para dos grupos distintos:[69]
- Pacientes con metástasis pulmonares aisladas (R2pulm) en el cuadro clínico inicial.
- Pacientes con tumores localizados que responden mal a la quimioterapia inicial (<90 % de necrosis) o con tumores de gran tamaño (>200 ml) (R2loc).
En ambos grupos de estudio se presentó la posibilidad de sesgo de selección de pacientes aptos para participar y que aceptaron someterse a la aleatorización, lo que limita la generalización de los resultados. Solo el 40 % de los pacientes aptos participó en la asignación aleatorizada.
- En los pacientes del grupo R2pulm, no hubo una diferencia estadísticamente significativa en la SSC y la SG según el grupo de tratamiento.[70]
- Las tasas de SSC a 3 años fueron del 50,6 % en los pacientes que recibieron VAI e irradiación pulmonar total versus un 56,6 % en los pacientes que recibieron BuMel. Las tasas de SSC a 8 años fueron del 43,1 % en los pacientes que recibieron VAI e irradiación pulmonar total versus un 52,9 % en los pacientes que recibieron BuMel.
- Las tasas de SG a 3 años fueron del 68 % en los pacientes que recibieron VAI con irradiación pulmonar total versus un 68,2 % en los pacientes que recibieron BuMel. Las tasas de SG a 8 años fueron del 54,2 % en los pacientes que recibieron VAI con irradiación pulmonar total versus un 55,3 % en los pacientes que recibieron BuMel.
- En el grupo de pacientes R2loc, la tasa de SSC a 3 años fue más alta en los que recibieron BuMel, en comparación con los que recibieron quimioterapia continuada (66,9 vs. 53,1 %; P = 0,019). La tasa de SG a 3 años fue del 78 % con BuMel y del 72,2 % con quimioterapia continuada (P = 0,028).[69]
- En los pacientes del grupo R2pulm, no hubo una diferencia estadísticamente significativa en la SSC y la SG según el grupo de tratamiento.[70]
El régimen de inducción que se utilizó en el ensayo EURO-EWING-INTERGROUP-EE99 fue VIDE, que incluye dosis menos intensas que el régimen de los estudios del COG. Esto se puede inferir de la intensidad de dosis prevista para los fármacos usados durante el período de 21 semanas previo a la aleatorización en el estudio EURO-EWING-INTERGROUP-EE99 (consultar el Cuadro 6). La menor intensidad de dosis también se puede inferir de los desenlaces del estudio EURO-EWING-INTERGROUP-EE99 en los pacientes clasificados en el estrato de enfermedad localizada. A continuación se presenta una síntesis de los resultados de este estudio:
- Los pacientes del estrato de menor riesgo, R1, eran pacientes con tumores primarios pequeños de volumen inferior a 200 ml. Además, los pacientes que presentaron una respuesta precaria durante los primeros 6 ciclos de VIDE, según la evaluación patológica o radiológica, pasaron del estrato R1 al R2. Esto llevó a que en el estrato R1 solo se incluyera a los pacientes con tumores primarios pequeños y respuesta favorable a la terapia inicial. La probabilidad de SSC a 3 años en este grupo de riesgo bajo (favorable) fue del 76 % y la tasa de SG a 3 años fue del 85 %.[28]
- En todos los pacientes con sarcoma de Ewing localizado, incluso los pacientes con tumores primarios grandes y pacientes con respuesta precaria al tratamiento inicial que participaron en el ensayo COG-AEWS1031 (NCT01231906), la probabilidad de SSC a 5 años fue del 73 % y la tasa de SG a 5 años fue del 88 %.[24]
La observación de que la terapia de dosis altas con rescate autógeno de células madre mejora los desenlaces de pacientes con respuesta precaria a la terapia inicial del estudio EURO-EWING-INTERGROUP-EE99 se debe interpretar en este contexto. La ventaja de la terapia de dosis altas como consolidación para pacientes con respuesta precaria al tratamiento inicial que recibieron un régimen menos intenso no se puede extrapolar a la población de pacientes que reciben un tratamiento más intensivo como terapia inicial.
Cuadro 6. Comparación de la intensidad de dosis del ensayo EURO-EWING-INTERGROUP-EE99 y la reducción del intervalo de dosis del COG Fármaco de quimioterapia Intensidad de dosis prescrita (mg/semana) Ensayo EURO-EWING-INTERGROUP-EE99 [28] Reducción del intervalo de dosis del COG [24] COG = Children's Oncology Group. Vincristina 0,5 mg/m2 0,43 mg/m2 Doxorrubicina 17,1 mg/m2 21,4 mg/m2 Ifosfamida 3000 mg/m2 2150 mg/m2 Ciclofosfamida 0 343 mg/m2 Dosis equivalente de ciclofosfamida (= dosis de ciclofosfamida + dosis de ifosfamida × 0,244) 732 mg/m2 868 mg/m2 - Se publicaron múltiples estudios pequeños que notificaron un beneficio del TCMH, pero cuya interpretación es difícil porque solo los pacientes con una buena respuesta inicial a la quimioterapia estándar se consideraron aptos para el trasplante.
Sarcoma de Ewing extraóseo
Desde el punto de vista biológico, el sarcoma de Ewing extraóseo es similar al sarcoma de Ewing que se origina en el hueso. Tradicionalmente, la mayoría de niños y adultos jóvenes con sarcoma de Ewing extraóseo se trataban con protocolos diseñados para el tratamiento del rabdomiosarcoma. Esto es importante porque muchos de los regímenes de tratamiento para el rabdomiosarcoma no incluyen una antraciclina, que es un componente fundamental de los regímenes actuales para el tratamiento del sarcoma de Ewing. En la actualidad, los pacientes con sarcoma de Ewing extraóseo son aptos para participar en estudios que incluyen el sarcoma de Ewing óseo.
Evidencia (tratamiento del sarcoma de Ewing extraóseo):
- De 1987 a 2004, 111 pacientes con sarcoma de Ewing extraóseo sin metástasis participaron en los protocolos RMS 88 y RMS-96.[71] Los pacientes sometidos a resección tumoral inicial completa recibieron ifosfamida, vincristina y actinomicina (IVA), mientras que los pacientes con tumor residual recibieron IVA con doxorrubicina (VAIA) o IVA con carboplatino, epirrubicina y etopósido (CEVAIE). El 76 % de los pacientes recibieron radiación.
- La tasa de SSC a 5 años fue del 59 %, y la tasa de SG fue del 69 %.
- En un análisis multivariante, los factores independientes de pronóstico adverso fueron tumor primario axial, tamaño del tumor mayor a 10 cm, clasificación en el Intergroup Rhabdomyosarcoma Studies Group III y ausencia de radioterapia.
- En un estudio retrospectivo francés, se trató a pacientes con sarcoma de Ewing extraóseo con un régimen para rabdomiosarcoma (sin antraciclinas) o un régimen para sarcoma de Ewing (con antraciclinas).[72,73]
- Los pacientes que recibieron el régimen con antraciclina tuvieron una SSC y SG significativamente mejores que quienes no recibieron antraciclinas.
- En dos ensayos de sarcoma de Ewing en América del Norte, se incluyeron pacientes con sarcoma de Ewing extraóseo.[24,74] En una revisión de los datos de los estudios POG-9354 (INT-0154) y EWS0031 (NCT00006734), se identificaron 213 pacientes con sarcoma de Ewing extraóseo y 826 pacientes con sarcoma de Ewing óseo.[75][Nivel de evidencia C2]
- El CRI de la SSC del sarcoma Ewing extraóseo fue más alto (0,62) y el sarcoma de Ewing extraóseo se consideró un factor de pronóstico favorable, independientemente de la edad, la raza y el sitio primario.
- Además de la revisión anterior, en el ensayo COG AEWS1031 (NCT01231906) se trató posteriormente a pacientes con un programa de quimioterapia de intervalos reducidos (cada 2 semanas). Los pacientes se asignaron al azar a recibir ciclos estándar de vincristina, doxorrubicina y ciclofosfamida que se alternaban con ifosfamida y etopósido o vincristina, topotecán y ciclofosfamida cada tercer ciclo. Hubo 116 pacientes con tumores extraóseos, 114 pacientes con tumores óseos pélvicos y 399 pacientes con tumores óseos no pélvicos.[26]
- No hubo diferencias en los desenlaces de los pacientes entre los regímenes de tratamiento.
- Las tasas de SSC a 5 años fueron del 75 % (IC 95 %, 65–82 %) en los pacientes con tumores óseos pélvicos, del 78 % (IC 95 %, 73–81 %) en los pacientes con tumores óseos fuera de la pelvis, y del 85 % (IC 95 %, 76–90 %) en los pacientes con sitios primarios extraóseos (valor de P general = 0,124).
- El Cooperative Weichteilsarkomstudiengruppe (CWS) llevó a cabo un análisis retrospectivo de 243 pacientes con sarcoma de Ewing extraóseo no metastásico tratados en 3 estudios consecutivos de sarcomas de tejidos blandos entre 1991 y 2008. Se usaron varios regímenes farmacológicos diferentes, todos con vincristina, doxorrubicina y alquilantes.[76]
- El desenlace mejoró con el tiempo, pero fue difícil asignar la causalidad a un tratamiento específico en estas comparaciones históricas.
- Los pacientes con tumores primarios de extremidades (en comparación con otros sitios) y los pacientes con tumores pequeños presentaron mejores desenlaces.
- La tasa de SSC a 5 años osciló entre el 57 % y el 79 %, el mejor desenlace se observó en el protocolo más reciente.
El sarcoma de Ewing cutáneo es un tumor de tejido blando que se presenta en la piel o el tejido subcutáneo y que se comporta como un tumor menos maligno que el sarcoma de Ewing óseo primario o el sarcoma de Ewing de tejido blando. Estos tumores pueden presentarse en todo el cuerpo, aunque las extremidades son los sitios más comunes, y casi siempre están localizados.
Evidencia (tratamiento del sarcoma de Ewing cutáneo):
- En una revisión de 78 casos notificados, algunos sin confirmación molecular, la tasa de SG fue del 91 %. El control local adecuado, definido como una resección completa con márgenes negativos, la radioterapia o un tratamiento combinado redujo significativamente la incidencia de recaída. En estos pacientes se suele usar la quimioterapia estándar indicada para el sarcoma de Ewing porque no hay datos que indiquen cuáles pacientes podrían recibir un tratamiento menos intensivo.[77,78]
- En una serie de 56 pacientes con sarcoma de Ewing cutáneo o subcutáneo, se confirmó el excelente desenlace que se obtiene con tratamiento sistémico estándar y control local. El intento de llevar a cabo una cirugía definitiva primaria a menudo conlleva la necesidad posterior de someter al paciente a radioterapia, cirugías adicionales o una cirugía con repercusiones sobre el funcionamiento, lo que respalda la recomendación de realizar solo una biopsia como procedimiento inicial, en lugar de una resección inicial no planificada.[79][Nivel de evidencia C2]
Bibliografía
- Fuchs B, Valenzuela RG, Sim FH: Pathologic fracture as a complication in the treatment of Ewing's sarcoma. Clin Orthop (415): 25-30, 2003. [PUBMED Abstract]
- Singh VM, Salunga RC, Huang VJ, et al.: Analysis of the effect of various decalcification agents on the quantity and quality of nucleic acid (DNA and RNA) recovered from bone biopsies. Ann Diagn Pathol 17 (4): 322-6, 2013. [PUBMED Abstract]
- Hoffer FA, Gianturco LE, Fletcher JA, et al.: Percutaneous biopsy of peripheral primitive neuroectodermal tumors and Ewing's sarcomas for cytogenetic analysis. AJR Am J Roentgenol 162 (5): 1141-2, 1994. [PUBMED Abstract]
- Craft A, Cotterill S, Malcolm A, et al.: Ifosfamide-containing chemotherapy in Ewing's sarcoma: The Second United Kingdom Children's Cancer Study Group and the Medical Research Council Ewing's Tumor Study. J Clin Oncol 16 (11): 3628-33, 1998. [PUBMED Abstract]
- Shankar AG, Pinkerton CR, Atra A, et al.: Local therapy and other factors influencing site of relapse in patients with localised Ewing's sarcoma. United Kingdom Children's Cancer Study Group (UKCCSG). Eur J Cancer 35 (12): 1698-704, 1999. [PUBMED Abstract]
- Nilbert M, Saeter G, Elomaa I, et al.: Ewing's sarcoma treatment in Scandinavia 1984-1990--ten-year results of the Scandinavian Sarcoma Group Protocol SSGIV. Acta Oncol 37 (4): 375-8, 1998. [PUBMED Abstract]
- Ferrari S, Mercuri M, Rosito P, et al.: Ifosfamide and actinomycin-D, added in the induction phase to vincristine, cyclophosphamide and doxorubicin, improve histologic response and prognosis in patients with non metastatic Ewing's sarcoma of the extremity. J Chemother 10 (6): 484-91, 1998. [PUBMED Abstract]
- Grier HE, Krailo MD, Tarbell NJ, et al.: Addition of ifosfamide and etoposide to standard chemotherapy for Ewing's sarcoma and primitive neuroectodermal tumor of bone. N Engl J Med 348 (8): 694-701, 2003. [PUBMED Abstract]
- Thacker MM, Temple HT, Scully SP: Current treatment for Ewing's sarcoma. Expert Rev Anticancer Ther 5 (2): 319-31, 2005. [PUBMED Abstract]
- Juergens C, Weston C, Lewis I, et al.: Safety assessment of intensive induction with vincristine, ifosfamide, doxorubicin, and etoposide (VIDE) in the treatment of Ewing tumors in the EURO-E.W.I.N.G. 99 clinical trial. Pediatr Blood Cancer 47 (1): 22-9, 2006. [PUBMED Abstract]
- Dunst J, Schuck A: Role of radiotherapy in Ewing tumors. Pediatr Blood Cancer 42 (5): 465-70, 2004. [PUBMED Abstract]
- Donaldson SS: Ewing sarcoma: radiation dose and target volume. Pediatr Blood Cancer 42 (5): 471-6, 2004. [PUBMED Abstract]
- Bacci G, Ferrari S, Longhi A, et al.: Role of surgery in local treatment of Ewing's sarcoma of the extremities in patients undergoing adjuvant and neoadjuvant chemotherapy. Oncol Rep 11 (1): 111-20, 2004. [PUBMED Abstract]
- Krasin MJ, Rodriguez-Galindo C, Davidoff AM, et al.: Efficacy of combined surgery and irradiation for localized Ewings sarcoma family of tumors. Pediatr Blood Cancer 43 (3): 229-36, 2004. [PUBMED Abstract]
- Bacci G, Longhi A, Briccoli A, et al.: The role of surgical margins in treatment of Ewing's sarcoma family tumors: experience of a single institution with 512 patients treated with adjuvant and neoadjuvant chemotherapy. Int J Radiat Oncol Biol Phys 65 (3): 766-72, 2006. [PUBMED Abstract]
- Paulussen M, Ahrens S, Burdach S, et al.: Primary metastatic (stage IV) Ewing tumor: survival analysis of 171 patients from the EICESS studies. European Intergroup Cooperative Ewing Sarcoma Studies. Ann Oncol 9 (3): 275-81, 1998. [PUBMED Abstract]
- Pinkerton CR, Bataillard A, Guillo S, et al.: Treatment strategies for metastatic Ewing's sarcoma. Eur J Cancer 37 (11): 1338-44, 2001. [PUBMED Abstract]
- Bernstein ML, Devidas M, Lafreniere D, et al.: Intensive therapy with growth factor support for patients with Ewing tumor metastatic at diagnosis: Pediatric Oncology Group/Children's Cancer Group Phase II Study 9457--a report from the Children's Oncology Group. J Clin Oncol 24 (1): 152-9, 2006. [PUBMED Abstract]
- Ladenstein R, Pötschger U, Le Deley MC, et al.: Primary disseminated multifocal Ewing sarcoma: results of the Euro-EWING 99 trial. J Clin Oncol 28 (20): 3284-91, 2010. [PUBMED Abstract]
- Miser JS, Krailo MD, Tarbell NJ, et al.: Treatment of metastatic Ewing's sarcoma or primitive neuroectodermal tumor of bone: evaluation of combination ifosfamide and etoposide--a Children's Cancer Group and Pediatric Oncology Group study. J Clin Oncol 22 (14): 2873-6, 2004. [PUBMED Abstract]
- Miser JS, Goldsby RE, Chen Z, et al.: Treatment of metastatic Ewing sarcoma/primitive neuroectodermal tumor of bone: evaluation of increasing the dose intensity of chemotherapy--a report from the Children's Oncology Group. Pediatr Blood Cancer 49 (7): 894-900, 2007. [PUBMED Abstract]
- Haeusler J, Ranft A, Boelling T, et al.: The value of local treatment in patients with primary, disseminated, multifocal Ewing sarcoma (PDMES). Cancer 116 (2): 443-50, 2010. [PUBMED Abstract]
- Paulussen M, Craft AW, Lewis I, et al.: Results of the EICESS-92 Study: two randomized trials of Ewing's sarcoma treatment--cyclophosphamide compared with ifosfamide in standard-risk patients and assessment of benefit of etoposide added to standard treatment in high-risk patients. J Clin Oncol 26 (27): 4385-93, 2008. [PUBMED Abstract]
- Womer RB, West DC, Krailo MD, et al.: Randomized controlled trial of interval-compressed chemotherapy for the treatment of localized Ewing sarcoma: a report from the Children's Oncology Group. J Clin Oncol 30 (33): 4148-54, 2012. [PUBMED Abstract]
- Cash T, Krailo MD, Buxton AB, et al.: Long-Term Outcomes in Patients With Localized Ewing Sarcoma Treated With Interval-Compressed Chemotherapy on Children's Oncology Group Study AEWS0031. J Clin Oncol 41 (30): 4724-4728, 2023. [PUBMED Abstract]
- Leavey PJ, Laack NN, Krailo MD, et al.: Phase III Trial Adding Vincristine-Topotecan-Cyclophosphamide to the Initial Treatment of Patients With Nonmetastatic Ewing Sarcoma: A Children's Oncology Group Report. J Clin Oncol 39 (36): 4029-4038, 2021. [PUBMED Abstract]
- Brennan B, Kirton L, Marec-Bérard P, et al.: Comparison of two chemotherapy regimens in patients with newly diagnosed Ewing sarcoma (EE2012): an open-label, randomised, phase 3 trial. Lancet 400 (10362): 1513-1521, 2022. [PUBMED Abstract]
- Le Deley MC, Paulussen M, Lewis I, et al.: Cyclophosphamide compared with ifosfamide in consolidation treatment of standard-risk Ewing sarcoma: results of the randomized noninferiority Euro-EWING99-R1 trial. J Clin Oncol 32 (23): 2440-8, 2014. [PUBMED Abstract]
- Brunetto AL, Castillo LA, Petrilli AS, et al.: Carboplatin in the treatment of Ewing sarcoma: Results of the first Brazilian collaborative study group for Ewing sarcoma family tumors-EWING1. Pediatr Blood Cancer 62 (10): 1747-53, 2015. [PUBMED Abstract]
- DuBois SG, Krailo MD, Gebhardt MC, et al.: Comparative evaluation of local control strategies in localized Ewing sarcoma of bone: a report from the Children's Oncology Group. Cancer 121 (3): 467-75, 2015. [PUBMED Abstract]
- Lin TA, Ludmir EB, Liao KP, et al.: Timing of Local Therapy Affects Survival in Ewing Sarcoma. Int J Radiat Oncol Biol Phys 104 (1): 127-136, 2019. [PUBMED Abstract]
- Yock TI, Krailo M, Fryer CJ, et al.: Local control in pelvic Ewing sarcoma: analysis from INT-0091--a report from the Children's Oncology Group. J Clin Oncol 24 (24): 3838-43, 2006. [PUBMED Abstract]
- Andreou D, Ranft A, Gosheger G, et al.: Which Factors Are Associated with Local Control and Survival of Patients with Localized Pelvic Ewing's Sarcoma? A Retrospective Analysis of Data from the Euro-EWING99 Trial. Clin Orthop Relat Res 478 (2): 290-302, 2020. [PUBMED Abstract]
- Foulon S, Brennan B, Gaspar N, et al.: Can postoperative radiotherapy be omitted in localised standard-risk Ewing sarcoma? An observational study of the Euro-E.W.I.N.G group. Eur J Cancer 61: 128-36, 2016. [PUBMED Abstract]
- Seitz G, Urla C, Sparber-Sauer M, et al.: Treatment and outcome of patients with thoracic tumors of the Ewing sarcoma family: A report from the Cooperative Weichteilsarkom Studiengruppe CWS-81, -86, -91, -96, and -2002P trials. Pediatr Blood Cancer 66 (3): e27537, 2019. [PUBMED Abstract]
- Shamberger RC, LaQuaglia MP, Gebhardt MC, et al.: Ewing sarcoma/primitive neuroectodermal tumor of the chest wall: impact of initial versus delayed resection on tumor margins, survival, and use of radiation therapy. Ann Surg 238 (4): 563-7; discussion 567-8, 2003. [PUBMED Abstract]
- Wagner TD, Kobayashi W, Dean S, et al.: Combination short-course preoperative irradiation, surgical resection, and reduced-field high-dose postoperative irradiation in the treatment of tumors involving the bone. Int J Radiat Oncol Biol Phys 73 (1): 259-66, 2009. [PUBMED Abstract]
- Hoffmann C, Ahrens S, Dunst J, et al.: Pelvic Ewing sarcoma: a retrospective analysis of 241 cases. Cancer 85 (4): 869-77, 1999. [PUBMED Abstract]
- Sucato DJ, Rougraff B, McGrath BE, et al.: Ewing's sarcoma of the pelvis. Long-term survival and functional outcome. Clin Orthop (373): 193-201, 2000. [PUBMED Abstract]
- Bacci G, Ferrari S, Mercuri M, et al.: Multimodal therapy for the treatment of nonmetastatic Ewing sarcoma of pelvis. J Pediatr Hematol Oncol 25 (2): 118-24, 2003. [PUBMED Abstract]
- Bacci G, Ferrari S, Longhi A, et al.: Local and systemic control in Ewing's sarcoma of the femur treated with chemotherapy, and locally by radiotherapy and/or surgery. J Bone Joint Surg Br 85 (1): 107-14, 2003. [PUBMED Abstract]
- Ozaki T, Hillmann A, Hoffmann C, et al.: Ewing's sarcoma of the femur. Prognosis in 69 patients treated by the CESS group. Acta Orthop Scand 68 (1): 20-4, 1997. [PUBMED Abstract]
- Ayoub KS, Fiorenza F, Grimer RJ, et al.: Extensible endoprostheses of the humerus after resection of bone tumours. J Bone Joint Surg Br 81 (3): 495-500, 1999. [PUBMED Abstract]
- Bacci G, Palmerini E, Staals EL, et al.: Ewing's sarcoma family tumors of the humerus: outcome of patients treated with radiotherapy, surgery or surgery and adjuvant radiotherapy. Radiother Oncol 93 (2): 383-7, 2009. [PUBMED Abstract]
- Casadei R, Magnani M, Biagini R, et al.: Prognostic factors in Ewing's sarcoma of the foot. Clin Orthop (420): 230-8, 2004. [PUBMED Abstract]
- Anakwenze OA, Parker WL, Wold LE, et al.: Ewing's sarcoma of the hand. J Hand Surg Eur Vol 34 (1): 35-9, 2009. [PUBMED Abstract]
- Shamberger RC, Laquaglia MP, Krailo MD, et al.: Ewing sarcoma of the rib: results of an intergroup study with analysis of outcome by timing of resection. J Thorac Cardiovasc Surg 119 (6): 1154-61, 2000. [PUBMED Abstract]
- Sirvent N, Kanold J, Levy C, et al.: Non-metastatic Ewing's sarcoma of the ribs: the French Society of Pediatric Oncology Experience. Eur J Cancer 38 (4): 561-7, 2002. [PUBMED Abstract]
- Schuck A, Ahrens S, Konarzewska A, et al.: Hemithorax irradiation for Ewing tumors of the chest wall. Int J Radiat Oncol Biol Phys 54 (3): 830-8, 2002. [PUBMED Abstract]
- Windfuhr JP: Primitive neuroectodermal tumor of the head and neck: incidence, diagnosis, and management. Ann Otol Rhinol Laryngol 113 (7): 533-43, 2004. [PUBMED Abstract]
- Venkateswaran L, Rodriguez-Galindo C, Merchant TE, et al.: Primary Ewing tumor of the vertebrae: clinical characteristics, prognostic factors, and outcome. Med Pediatr Oncol 37 (1): 30-5, 2001. [PUBMED Abstract]
- Marco RA, Gentry JB, Rhines LD, et al.: Ewing's sarcoma of the mobile spine. Spine 30 (7): 769-73, 2005. [PUBMED Abstract]
- Schuck A, Ahrens S, von Schorlemer I, et al.: Radiotherapy in Ewing tumors of the vertebrae: treatment results and local relapse analysis of the CESS 81/86 and EICESS 92 trials. Int J Radiat Oncol Biol Phys 63 (5): 1562-7, 2005. [PUBMED Abstract]
- Bacci G, Boriani S, Balladelli A, et al.: Treatment of nonmetastatic Ewing's sarcoma family tumors of the spine and sacrum: the experience from a single institution. Eur Spine J 18 (8): 1091-5, 2009. [PUBMED Abstract]
- Kushner BH, Meyers PA: How effective is dose-intensive/myeloablative therapy against Ewing's sarcoma/primitive neuroectodermal tumor metastatic to bone or bone marrow? The Memorial Sloan-Kettering experience and a literature review. J Clin Oncol 19 (3): 870-80, 2001. [PUBMED Abstract]
- Marina N, Meyers PA: High-dose therapy and stem-cell rescue for Ewing's family of tumors in second remission. J Clin Oncol 23 (19): 4262-4, 2005. [PUBMED Abstract]
- Burdach S: Treatment of advanced Ewing tumors by combined radiochemotherapy and engineered cellular transplants. Pediatr Transplant 8 (Suppl 5): 67-82, 2004. [PUBMED Abstract]
- McTiernan A, Driver D, Michelagnoli MP, et al.: High dose chemotherapy with bone marrow or peripheral stem cell rescue is an effective treatment option for patients with relapsed or progressive Ewing's sarcoma family of tumours. Ann Oncol 17 (8): 1301-5, 2006. [PUBMED Abstract]
- Burdach S, Meyer-Bahlburg A, Laws HJ, et al.: High-dose therapy for patients with primary multifocal and early relapsed Ewing's tumors: results of two consecutive regimens assessing the role of total-body irradiation. J Clin Oncol 21 (16): 3072-8, 2003. [PUBMED Abstract]
- Meyers PA, Krailo MD, Ladanyi M, et al.: High-dose melphalan, etoposide, total-body irradiation, and autologous stem-cell reconstitution as consolidation therapy for high-risk Ewing's sarcoma does not improve prognosis. J Clin Oncol 19 (11): 2812-20, 2001. [PUBMED Abstract]
- Oberlin O, Rey A, Desfachelles AS, et al.: Impact of high-dose busulfan plus melphalan as consolidation in metastatic Ewing tumors: a study by the Société Française des Cancers de l'Enfant. J Clin Oncol 24 (24): 3997-4002, 2006. [PUBMED Abstract]
- Hawkins D, Barnett T, Bensinger W, et al.: Busulfan, melphalan, and thiotepa with or without total marrow irradiation with hematopoietic stem cell rescue for poor-risk Ewing-Sarcoma-Family tumors. Med Pediatr Oncol 34 (5): 328-37, 2000. [PUBMED Abstract]
- Rosenthal J, Bolotin E, Shakhnovits M, et al.: High-dose therapy with hematopoietic stem cell rescue in patients with poor prognosis Ewing family tumors. Bone Marrow Transplant 42 (5): 311-8, 2008. [PUBMED Abstract]
- Burdach S, Thiel U, Schöniger M, et al.: Total body MRI-governed involved compartment irradiation combined with high-dose chemotherapy and stem cell rescue improves long-term survival in Ewing tumor patients with multiple primary bone metastases. Bone Marrow Transplant 45 (3): 483-9, 2010. [PUBMED Abstract]
- Gaspar N, Rey A, Bérard PM, et al.: Risk adapted chemotherapy for localised Ewing's sarcoma of bone: the French EW93 study. Eur J Cancer 48 (9): 1376-85, 2012. [PUBMED Abstract]
- Drabko K, Raciborska A, Bilska K, et al.: Consolidation of first-line therapy with busulphan and melphalan, and autologous stem cell rescue in children with Ewing's sarcoma. Bone Marrow Transplant 47 (12): 1530-4, 2012. [PUBMED Abstract]
- Loschi S, Dufour C, Oberlin O, et al.: Tandem high-dose chemotherapy strategy as first-line treatment of primary disseminated multifocal Ewing sarcomas in children, adolescents and young adults. Bone Marrow Transplant 50 (8): 1083-8, 2015. [PUBMED Abstract]
- Thiel U, Wawer A, Wolf P, et al.: No improvement of survival with reduced- versus high-intensity conditioning for allogeneic stem cell transplants in Ewing tumor patients. Ann Oncol 22 (7): 1614-21, 2011. [PUBMED Abstract]
- Whelan J, Le Deley MC, Dirksen U, et al.: High-Dose Chemotherapy and Blood Autologous Stem-Cell Rescue Compared With Standard Chemotherapy in Localized High-Risk Ewing Sarcoma: Results of Euro-E.W.I.N.G.99 and Ewing-2008. J Clin Oncol : JCO2018782516, 2018. [PUBMED Abstract]
- Dirksen U, Brennan B, Le Deley MC, et al.: High-Dose Chemotherapy Compared With Standard Chemotherapy and Lung Radiation in Ewing Sarcoma With Pulmonary Metastases: Results of the European Ewing Tumour Working Initiative of National Groups, 99 Trial and EWING 2008. J Clin Oncol 37 (34): 3192-3202, 2019. [PUBMED Abstract]
- Spiller M, Bisogno G, Ferrari A, et al.: Prognostic factors in localized extraosseus Ewing family tumors. [Abstract] Pediatr Blood Cancer 46 (10) : A-PD.024, 434, 2006.
- Castex MP, Rubie H, Stevens MC, et al.: Extraosseous localized ewing tumors: improved outcome with anthracyclines--the French society of pediatric oncology and international society of pediatric oncology. J Clin Oncol 25 (10): 1176-82, 2007. [PUBMED Abstract]
- Dantonello TM, Int-Veen C, Harms D, et al.: Cooperative trial CWS-91 for localized soft tissue sarcoma in children, adolescents, and young adults. J Clin Oncol 27 (9): 1446-55, 2009. [PUBMED Abstract]
- Granowetter L, Womer R, Devidas M, et al.: Dose-intensified compared with standard chemotherapy for nonmetastatic Ewing sarcoma family of tumors: a Children's Oncology Group Study. J Clin Oncol 27 (15): 2536-41, 2009. [PUBMED Abstract]
- Cash T, McIlvaine E, Krailo MD, et al.: Comparison of clinical features and outcomes in patients with extraskeletal versus skeletal localized Ewing sarcoma: A report from the Children's Oncology Group. Pediatr Blood Cancer 63 (10): 1771-9, 2016. [PUBMED Abstract]
- Koscielniak E, Sparber-Sauer M, Scheer M, et al.: Extraskeletal Ewing sarcoma in children, adolescents, and young adults. An analysis of three prospective studies of the Cooperative Weichteilsarkomstudiengruppe (CWS). Pediatr Blood Cancer 68 (10): e29145, 2021. [PUBMED Abstract]
- Collier AB, Simpson L, Monteleone P: Cutaneous Ewing sarcoma: report of 2 cases and literature review of presentation, treatment, and outcome of 76 other reported cases. J Pediatr Hematol Oncol 33 (8): 631-4, 2011. [PUBMED Abstract]
- Terrier-Lacombe MJ, Guillou L, Chibon F, et al.: Superficial primitive Ewing's sarcoma: a clinicopathologic and molecular cytogenetic analysis of 14 cases. Mod Pathol 22 (1): 87-94, 2009. [PUBMED Abstract]
- Di Giannatale A, Frezza AM, Le Deley MC, et al.: Primary cutaneous and subcutaneous Ewing sarcoma. Pediatr Blood Cancer 62 (9): 1555-61, 2015. [PUBMED Abstract]
Consideraciones especiales para el tratamiento de niños con cáncer
El cáncer en niños y adolescentes es raro, aunque desde 1975 se ha observado un aumento gradual de la incidencia general.[1] Los niños y adolescentes con cáncer se deben derivar a centros médicos que cuenten con equipos multidisciplinarios de especialistas en oncología con experiencia en el tratamiento de los cánceres que se presentan en la niñez y la adolescencia. Este equipo multidisciplinario incorpora la pericia de los siguientes profesionales de atención de la salud y otros para asegurar que los niños reciban el tratamiento, los cuidados médicos de apoyo y la rehabilitación que les permitan lograr una supervivencia y calidad de vida óptimas:
- Médicos de atención primaria.
- Cirujanos pediatra.
- Cirujanos de trasplante.
- Patólogos.
- Radioncólogos pediatras.
- Oncólogos y hematólogos pediatras.
- Oftalmólogos.
- Especialistas en rehabilitación.
- Enfermeros especializados en oncología pediátrica.
- Trabajadores o asistentes sociales.
- Profesionales de la vida infantil.
- Psicólogos.
- Nutricionistas y dietistas.
Para obtener información específica sobre los cuidados médicos de apoyo para niños y adolescentes con cáncer, consultar los resúmenes de Cuidados médicos de apoyo y cuidados paliativos.
La American Academy of Pediatrics estableció pautas para los centros de oncología pediátrica y su función en el tratamiento de los pacientes con cáncer infantil.[2] En estos centros de oncología pediátrica, se dispone de ensayos clínicos para la mayoría de los tipos de cáncer que se presentan en niños y adolescentes, y se ofrece la oportunidad de participar a la mayoría de los pacientes y familiares. Por lo general, los ensayos clínicos para los niños y adolescentes con cáncer se diseñan a fin de comparar un tratamiento que parece mejor con el tratamiento estándar actual. La mayoría de los avances en la identificación de tratamientos curativos para los cánceres infantiles se lograron mediante ensayos clínicos. Para obtener información sobre los ensayos clínicos en curso, consultar el portal de Internet del NCI.
Los niños y adolescentes sobrevivientes de cáncer necesitan un seguimiento minucioso, ya que es posible que los efectos secundarios del tratamiento del cáncer persistan, o se presenten, meses o años después de este. Para obtener información específica sobre la incidencia, el tipo y la vigilancia de los efectos tardíos en los niños y adolescentes sobrevivientes de cáncer, consultar Efectos tardíos del tratamiento anticanceroso en la niñez.
Bibliografía
- Smith MA, Seibel NL, Altekruse SF, et al.: Outcomes for children and adolescents with cancer: challenges for the twenty-first century. J Clin Oncol 28 (15): 2625-34, 2010. [PUBMED Abstract]
- American Academy of Pediatrics: Standards for pediatric cancer centers. Pediatrics 134 (2): 410-4, 2014. Also available online. Last accessed February 25, 2025.
Tratamiento del sarcoma de Ewing localizado
Las opciones de tratamiento estándar para el sarcoma de Ewing localizado son las siguientes:
Dado que la mayoría de los pacientes con enfermedad aparentemente localizada en el momento del diagnóstico tienen enfermedad metastásica oculta, la quimioterapia multifarmacológica y la cirugía o radioterapia (para controlar la enfermedad local) están indicadas en el tratamiento de todos los pacientes.[1-8] Los pacientes con sarcoma de Ewing localizado que reciben regímenes de tratamiento actuales logran tasas de supervivencia sin complicaciones (SSC) y supervivencia general (SG) de cerca del 70 % a los 5 años del diagnóstico.[9]
Quimioterapia
La quimioterapia estándar vigente en los Estados Unidos incluye vincristina, doxorrubicina y ciclofosfamida (VDC), en alternancia con ifosfamida y etopósido (IE) o VDC/IE.[9]; [10][Nivel de evidencia A1] A partir de los resultados del ensayo EURO EWING 2012 (EE2012), en el que se comparó VDC/IE con VIDE (vincristina, ifosfamida, doxorrubicina y etopósido), el uso del régimen VDC/IE a nivel internacional como tratamiento inicial, en lugar del anterior régimen VIDE, ha aumentado.[11][Nivel de evidencia B1] Para obtener más información sobre el ensayo EE2012, consultar la sección Quimioterapia para el sarcoma de Ewing.
Evidencia (quimioterapia):
- IE ha demostrado actividad en el sarcoma de Ewing. En un gran ensayo clínico aleatorizado y en un ensayo no aleatorizado, se demostró mejora del desenlace cuando se administró IE en alternancia con VDC.[9,12]
- La utilización de dosis altas de VDC mostró resultados promisorios en un número pequeño de pacientes. En un estudio de una sola institución de 44 pacientes tratados con dosis altas de VDC e IE, se observó una tasa de SSC a 4 años del 82 %.[13]
- Sin embargo, en un ensayo intergrupal del Pediatric Oncology Group y el Children's Cancer Group, en el que se comparó un régimen de quimioterapia con dosis intensivas de VDC/IE y alquilantes con un régimen de dosis estándar de los mismos fármacos no se observaron diferencias en los desenlaces.[14] A diferencia del ensayo de una sola institución, en este ensayo no se mantuvo la intensidad de la dosis de ciclofosfamida durante la duración del tratamiento.[13]
- En un ensayo del Children's Oncology Group (COG) (COG-AEWS0031 [NCT00006734]), 568 pacientes con sarcoma de Ewing extradural localizado recién diagnosticado se asignaron al azar para recibir quimioterapia (VDC/IE) administrado cada 2 semanas (intervalo reducido) o cada 3 semanas (estándar).[10]
- Los pacientes asignados al azar al intervalo de tratamiento de 2 semanas presentaron una mejora de la tasa de SSC a 5 años (73 vs. 65 %, P = 0,048).
- No se observó aumento de la toxicidad en el programa de cada 2 semanas.
- Al cabo de un seguimiento más prolongado de 10 años, se continuó observando una SSC superior con el régimen de VDC/IE de intervalo reducido. La SG también fue significativamente más alta con este régimen que con el de VDC/IE administrado cada 3 semanas.[15]
- En el ensayo European Ewing 2008R1 se evaluaron 284 pacientes con sarcoma de Ewing de riesgo estándar, definido como enfermedad localizada con respuesta histológica favorable a la quimioterapia inicial o volumen tumoral inicial de 200 ml o menos. Todos los pacientes recibieron quimioterapia VIDE seguida de terapia de consolidación con vincristina, dactinomicina e ifosfamida (VAI) (hombres) o vincristina, dactinomicina y ciclofosfamida (VAC) (mujeres). En el sexto ciclo de consolidación, los pacientes se asignaron al azar a un grupo que recibió terapia con adición de ácido zoledrónico durante 9 ciclos de 28 días o a un grupo que no recibió terapia de mantenimiento.[16]
- Los desenlaces fueron similares entre los pacientes que recibieron ácido zoledrónico y los que no recibieron terapia de mantenimiento (tasas de SSC a 3 años, 84 % vs. 81,7 %).
- Los pacientes que recibieron ácido zoledrónico presentaron tasas más altas de efectos tóxicos renales, neurológicos y gastrointestinales.
- El EE2012 fue un ensayo de fase III, controlado, aleatorizado, sin enmascaramiento que iniciaron investigadores europeos y se realizó en 10 países. Entre 2014 y 2019, se incluyeron 640 pacientes que se asignaron al azar (1:1) a los regímenes de tratamiento europeo o del COG (320 pacientes en cada grupo de tratamiento). El régimen de tratamiento europeo para la inducción incluyó VIDE, y el régimen de consolidación incluyó vincristina, dactinomicina, con ifosfamida o ciclofosfamida, o busulfano y melfalán (grupo 1). El régimen de tratamiento del COG para la inducción incluyó VDC/IE, y el régimen de consolidación incluyó vincristina y ciclofosfamida, con ifosfamida y etopósido o busulfano y melfalán (grupo 2).[11][Nivel de evidencia B1]
- La tasa de SSC a 3 años fue del 61 % en los pacientes del grupo 1 y del 67 % en los pacientes del grupo 2 (cociente de riesgos instantáneos ajustado [CRI], 0,71; intervalo de confianza [IC] 95 %, 0,55–0,92 que favoreció al grupo 2).
- La probabilidad de que el CRI real para el grupo 2 con VDC/IE de intervalos reducidos fuera inferior a 1 fue mayor de 0,99.
- Los investigadores llegaron a la conclusión de que la quimioterapia de inducción de dosis intensiva con el régimen VDC/IE era más eficaz, menos tóxica, y más breve para pacientes en todos los estadios de sarcoma de Ewing recién diagnosticado que el régimen VIDE.
Medidas de control local
El control local se puede lograr mediante cirugía o radioterapia. Las decisiones sobre la modalidad ideal para el control local de un paciente implica analizar los siguientes aspectos:
- La posibilidad de resección completa con márgenes adecuados después de un período inicial de tratamiento sistémico.
- La repercusión funcional anticipada de un procedimiento quirúrgico.
- Las complicaciones previstas de la radioterapia.
- La posibilidad de que aumente el riesgo de segundas neoplasias malignas después de la radioterapia.
En un análisis con puntaje de propensión (método para ajustar según el sesgo de selección inherente a la ubicación y tamaño del tumor) para ajustar según las características clínicas que pudieran afectar la preferencia por cirugía sola, radiación sola o cirugía y radioterapia combinadas se demostró que se obtienen tasas similares de SSC con cada método de terapia local después del ajuste por propensión.[17]
Cirugía
Por lo general, la cirugía es el abordaje preferido si la lesión es resecable.[18,19] No se ha probado en ningún ensayo aleatorizado prospectivo la superioridad de la resección para el control local. La aparente superioridad quizás represente un sesgo de selección.
- En estudios pasados, los tumores más pequeños y de ubicación más periférica casi siempre se trataban con cirugía, y los tumores más grandes y de ubicación más central casi siempre se trataban con radioterapia.[20]
- En un estudio retrospectivo italiano, se observó que la cirugía solo mejoró los resultados en los tumores de extremidades, aunque el número de pacientes con sarcoma de Ewing del eje central en quienes se lograron márgenes adecuados fue pequeño.[8]
- En una serie de 39 pacientes sometidos a cirugía y radioterapia en el St. Jude Children’s Research Hospital, la tasa de fracaso local a 8 años fue del 5 % en los pacientes con márgenes quirúrgicos negativos y del 17 % en aquellos con márgenes positivos.[5]
- Los datos de pacientes con sarcoma de Ewing primario en la pelvis en un ensayo intergrupal norteamericano no mostraron diferencias en el control local o la supervivencia según la modalidad de control (cirugía sola, radioterapia sola o radiación con cirugía).[21]
- Los pacientes con un tumor residual viable en la pieza resecada tienen un desenlace más precario que aquellos que exhiben una necrosis completa. En un estudio francés sobre sarcoma de Ewing (EW88), las tasas de SSC fueron del 75 % en los pacientes con menos de un 5% de tumor viable, del 48 % en los pacientes con un 5 % a un 30 % de tumor viable y del 20 % en los pacientes con más de un 30 % de tumor viable.[20]
En un análisis retrospectivo de una sola institución de 78 pacientes con sarcoma de Ewing, se indicó que la fractura patológica en el momento de la presentación inicial se relacionó con una SSC y SG inferiores.[22][Nivel de evidencia C1] En otro estudio se encontró que la fractura patológica en el momento del diagnóstico no impedía la resección quirúrgica y no se relacionaba con un desenlace adverso.[23]
Radioterapia
La radioterapia suele administrarse en los siguientes casos:
- Pacientes que no tienen la opción de una cirugía que conserve la funcionalidad y el aspecto estético.
- Pacientes cuyos tumores se extirparon, pero con márgenes inadecuados.
- Pacientes que pueden recibir radioterapia prequirúrgica si es posible una resección macroscópica total, pero sin márgenes adecuados (y conservación de la funcionalidad y el aspecto estético).
La radioterapia se administra en un entorno en el que se aplican técnicas estrictas de planificación por profesionales experimentados en el tratamiento del sarcoma de Ewing. Este abordaje dará lugar a control local del tumor con morbilidad aceptable en la mayoría de los pacientes.[1,2,24]
La dosis de radiación se puede ajustar según el grado de enfermedad residual después del procedimiento quirúrgico inicial. Cuando no se realiza una resección quirúrgica, por lo general se administra la radioterapia en dosis fraccionadas, por un total de casi 55,8 Gy dirigidos al volumen tumoral previo a la quimioterapia. En un estudio aleatorizado de 40 pacientes con sarcoma de Ewing en el que se usaron 55,8 Gy dirigidos a la zona que ocupaba el tumor antes de la quimioterapia, con un margen de 2 cm, se comparó este tratamiento con la misma dosis tumoral luego de 39,6 Gy dirigidos al hueso entero y no se observó diferencia en el control local ni la SSC.[3] La radioterapia hiperfraccionada no se relacionó con un mejor control local ni una disminución de la morbilidad.[1]
La radioterapia preoperatoria es un abordaje que a veces se usa cuando se puede hacer una resección quirúrgica, pero con probabilidad de enfermedad residual microscópica. Un grupo de expertos internacionales usó un método de Delphi de 3 etapas modificado para formular declaraciones de consenso sobre el tratamiento local. El grupo llegó a un consenso sólido sobre la administración preoperatoria de radioterapia cuando en las imágenes se anticipa que alguno de los márgenes será inadecuado en la resección.[25]
En los pacientes con enfermedad residual después de un intento de resección quirúrgica, el Intergroup Ewing Sarcoma Study (INT-0091) recomienda administrar 45 Gy dirigidos al sitio original de la enfermedad con un refuerzo de 10,8 Gy en los pacientes con enfermedad residual macroscópica, y 45 Gy con un refuerzo de 5,4 Gy en los pacientes con enfermedad residual microscópica. No se recomienda administrar radioterapia a los pacientes que no muestran indicios de enfermedad residual microscópica después de una resección quirúrgica.[14]
Se administra radioterapia a los pacientes que tienen una enfermedad irresecable después de la quimioterapia de inducción; se usan las mismas dosis que para los pacientes sometidos a resección parcial.[26] Por lo general, los pacientes con enfermedad irresecable son aquellos con tumores en las extremidades que tienen envoltura persistente de los haces neurovasculares o escisión quirúrgica mórbida que conlleva pérdida de funcionalidad. En un ensayo clínico controlado aleatorizado de fase III de pacientes con enfermedad irresecable, los pacientes se asignaron al azar a recibir radioterapia de dosis estándar (55,8 Gy en fracciones de 1,8 Gy) o radioterapia de dosis escalonadas (70,2 Gy en fracciones de 1,8 Gy).[26] Entre 2005 y 2015, en el estudio se incluyeron 47 pacientes que recibieron radioterapia de dosis estándar y 48 pacientes que recibieron radioterapia de dosis aumentadas (edad intercuartílica, 13–23 años). La mediana de la mayor dimensión del tumor fue de 9,7 cm. Al cabo de una mediana de seguimiento de hasta 67 meses, la tasa de control local a 5 años fue significativamente más alta en el grupo de dosis escalonadas que en el grupo de dosis estándar (76,4 vs. 49,4 %; P = 0,02). Las diferencias en las tasas de supervivencia sin enfermedad (SSE) y SG a los 5 años no fueron estadísticamente significativas (tasas de SSE, 46,7 vs. 31,8 %; P = 0,22; tasas de SG, 58,8 vs. 45,4 %; P = 0,08), posiblemente porque la tasa de enfermedad metastásica no cambió. La toxicidad cutánea de grado 2 o más fue mayor en el grupo de dosis altas (10,4 vs. 2,1 %; P = 0,08).
En un estudio no aleatorizado de una sola institución, los pacientes con tumores primarios de 8 cm o más, recibieron dosis más altas de radioterapia (mediana de dosis, 64,8 Gy). La incidencia acumulada a 5 años de la tasa de fracaso local fue del 6,6 %, que se compara de manera favorable con otras tasas de fracaso locales publicadas para este grupo de pacientes.[27]
En la comparación entre planes de tratamiento con radioterapia de haz de protones y con radioterapia de intensidad modulada (RTIM), se observó que la radioterapia de haz de protones puede conservar más tejido normal adyacente a los tumores primarios del sarcoma de Ewing que la RTIM.[28] El seguimiento hasta el momento es relativamente corto y no hay datos para determinar si una reducción en la dosis dirigida al tejido adyacente mejoraría los resultados funcionales o reduciría el riesgo de una segunda neoplasia maligna. Debido a que el número de pacientes es pequeño y el seguimiento es relativamente corto, no es posible determinar si el riesgo de recidiva local aumentaría al reducir la dosis de radiación dirigida a los tejidos adyacentes al tumor primario.
Se observaron tasas más altas de fracaso local en pacientes mayores de 14 años que presentaban tumores de más de 8 cm de longitud.[29] En los pacientes con tumores pélvicos, presentar un volumen tumoral más grande, un sitio tumoral periacetabular y el uso de radioterapia definitiva sola (en lugar de un abordaje de modalidad combinada) se relacionaron con tasas más altas de fracaso local.[30] En un análisis retrospectivo de pacientes con sarcoma de Ewing de pared torácica, se comparó a los pacientes que recibieron radioterapia dirigida al hemitórax con aquellos que recibieron radioterapia dirigida solo a la pared torácica. Los pacientes con invasión pleural, derrame pleural o contaminación intraoperatoria se asignaron a radioterapia dirigida a un hemitórax. La SSC fue más prolongada en los pacientes que recibieron radiación dirigida a un hemitórax, pero la diferencia no fue estadísticamente significativa. Además, la mayoría de pacientes con tumores vertebrales primarios no recibieron radiación dirigida al hemitórax y tuvieron una probabilidad más baja de SSC.[31]
La radioterapia se relaciona con neoplasias malignas subsiguientes. En un estudio retrospectivo, se observó que los pacientes que recibieron 60 Gy o más de radiación presentaron una incidencia de segundas neoplasias malignas secundarias del 20 %. Quienes recibieron una dosis de entre 48 Gy y 60 Gy tuvieron una incidencia del 5 % y quienes recibieron menos de 48 Gy no presentaron segundas neoplasia malignas.[32]
Dosis altas de quimioterapia con rescate autógeno de células madre
Evidencia (quimioterapia de dosis alta y rescate autógeno de células madre):
- La función de la terapia con dosis altas de busulfano y melfalán (BuMel) seguida de rescate de células madre se investigó en el ensayo prospectivo, aleatorizado EURO-EWING-INTERGROUP-EE99 (NCT00020566) para dos grupos distintos:[33]
- Pacientes con metástasis pulmonares aisladas (R2pulm) en el cuadro clínico inicial.
- Pacientes con tumores localizados que responden mal a la quimioterapia inicial (<90 % de necrosis) o con tumores de gran tamaño (>200 ml) (R2loc).
En ambos grupos de estudio se presentó la posibilidad de sesgo de selección de pacientes aptos para participar y que aceptaron someterse a la aleatorización, lo que limita la generalización de los resultados. Solo el 40 % de los pacientes aptos participó en la asignación aleatorizada.
- En los pacientes del grupo R2pulm, no hubo una diferencia estadísticamente significativa en la SSC o la SG según el grupo de tratamiento.[34]
- La tasa de SSC a 3 años fue del 50,6 % en los pacientes que recibieron vincristina, dactinomicina e ifosfamida (VAI) con irradiación pulmonar total versus un 56,6 % en los pacientes que recibieron BuMel.
- Las tasas de SSC a 8 años fueron del 43,1 % en los pacientes que recibieron VAI e irradiación pulmonar total versus un 52,9 % en los pacientes que recibieron BuMel.
- Las tasas de SG a 3 años fueron del 68 % en los pacientes que recibieron VAI con irradiación pulmonar total versus un 68,2 % en los pacientes que recibieron BuMel.
- Las tasas de SG a 8 años fueron del 54,2 % en los pacientes que recibieron VAI con irradiación pulmonar total versus un 55,3 % en los pacientes que recibieron BuMel.
- En el grupo de pacientes R2loc, la tasa de SSC a 3 años fue más alta en los que recibieron BuMel, en comparación con los que recibieron quimioterapia continuada (66,9 vs. 53,1 %; P = 0,019). La tasa de SG a 3 años fue del 78 % con BuMel y del 72,2 % con quimioterapia continuada (P = 0,028).[33]
- En los pacientes del grupo R2pulm, no hubo una diferencia estadísticamente significativa en la SSC o la SG según el grupo de tratamiento.[34]
El régimen de inducción que se utilizó en el ensayo EURO-EWING-INTERGROUP-EE99 fue VIDE, que incluye dosis menos intensas que el régimen de los estudios del COG. Esto se puede inferir de la intensidad de dosis prevista para los fármacos usados durante el período de 21 semanas previo a la aleatorización en el estudio EURO-EWING-INTERGROUP-EE99 (consultar el Cuadro 6 La menor intensidad de dosis también se puede inferir de los desenlaces del estudio EURO-EWING-INTERGROUP-EE99 en los pacientes clasificados en el estrato de enfermedad localizada. A continuación se presenta una síntesis de los resultados de este estudio:
- Los pacientes del estrato de menor riesgo, R1, eran pacientes con tumores primarios pequeños de volumen inferior a 200 ml. Además, los pacientes que presentaron una respuesta precaria durante los primeros 6 ciclos de VIDE, según la evaluación patológica o radiológica, pasaron del estrato R1 al R2. Esto llevó a que en el estrato R1 solo se incluyera a los pacientes con tumores primarios pequeños y respuesta favorable a la terapia inicial.[35]
- La probabilidad de SSC a 3 años en este grupo de riesgo bajo (favorable) fue del 76 % y la tasa de SG a 3 años fue del 85 %.
- En todos los pacientes con sarcoma de Ewing localizado, incluso los pacientes con tumores primarios grandes y pacientes con respuesta precaria al tratamiento inicial que participaron en el ensayo COG-AEWS1031 (NCT01231906), la tasa de supervivencia sin complicaciones a 5 años fue del 73 % y la tasa de SG a 5 años fue del 88 %.[10]
La observación de que la terapia de dosis altas con rescate autógeno de células madre mejora los desenlaces de pacientes con respuesta precaria a la terapia inicial del estudio EURO-EWING-INTERGROUP-EE99 se debe interpretar en este contexto. La ventaja de la terapia de dosis altas como consolidación para pacientes con respuesta precaria al tratamiento inicial que recibieron un régimen menos intenso no se puede extrapolar a la población de pacientes que reciben un tratamiento más intensivo como terapia inicial.
Ensayos clínicos en curso
Realizar una búsqueda avanzada en inglés de los ensayos clínicos sobre cáncer auspiciados por el NCI que ahora aceptan pacientes. La búsqueda se puede simplificar por ubicación del ensayo, tipo de tratamiento, nombre del fármaco y otros criterios. También se dispone de información general sobre los ensayos clínicos.
Bibliografía
- Dunst J, Jürgens H, Sauer R, et al.: Radiation therapy in Ewing's sarcoma: an update of the CESS 86 trial. Int J Radiat Oncol Biol Phys 32 (4): 919-30, 1995. [PUBMED Abstract]
- Donaldson SS, Torrey M, Link MP, et al.: A multidisciplinary study investigating radiotherapy in Ewing's sarcoma: end results of POG #8346. Pediatric Oncology Group. Int J Radiat Oncol Biol Phys 42 (1): 125-35, 1998. [PUBMED Abstract]
- Craft A, Cotterill S, Malcolm A, et al.: Ifosfamide-containing chemotherapy in Ewing's sarcoma: The Second United Kingdom Children's Cancer Study Group and the Medical Research Council Ewing's Tumor Study. J Clin Oncol 16 (11): 3628-33, 1998. [PUBMED Abstract]
- Nilbert M, Saeter G, Elomaa I, et al.: Ewing's sarcoma treatment in Scandinavia 1984-1990--ten-year results of the Scandinavian Sarcoma Group Protocol SSGIV. Acta Oncol 37 (4): 375-8, 1998. [PUBMED Abstract]
- Krasin MJ, Davidoff AM, Rodriguez-Galindo C, et al.: Definitive surgery and multiagent systemic therapy for patients with localized Ewing sarcoma family of tumors: local outcome and prognostic factors. Cancer 104 (2): 367-73, 2005. [PUBMED Abstract]
- Bacci G, Forni C, Longhi A, et al.: Long-term outcome for patients with non-metastatic Ewing's sarcoma treated with adjuvant and neoadjuvant chemotherapies. 402 patients treated at Rizzoli between 1972 and 1992. Eur J Cancer 40 (1): 73-83, 2004. [PUBMED Abstract]
- Rosito P, Mancini AF, Rondelli R, et al.: Italian Cooperative Study for the treatment of children and young adults with localized Ewing sarcoma of bone: a preliminary report of 6 years of experience. Cancer 86 (3): 421-8, 1999. [PUBMED Abstract]
- Bacci G, Longhi A, Briccoli A, et al.: The role of surgical margins in treatment of Ewing's sarcoma family tumors: experience of a single institution with 512 patients treated with adjuvant and neoadjuvant chemotherapy. Int J Radiat Oncol Biol Phys 65 (3): 766-72, 2006. [PUBMED Abstract]
- Grier HE, Krailo MD, Tarbell NJ, et al.: Addition of ifosfamide and etoposide to standard chemotherapy for Ewing's sarcoma and primitive neuroectodermal tumor of bone. N Engl J Med 348 (8): 694-701, 2003. [PUBMED Abstract]
- Womer RB, West DC, Krailo MD, et al.: Randomized controlled trial of interval-compressed chemotherapy for the treatment of localized Ewing sarcoma: a report from the Children's Oncology Group. J Clin Oncol 30 (33): 4148-54, 2012. [PUBMED Abstract]
- Brennan B, Kirton L, Marec-Bérard P, et al.: Comparison of two chemotherapy regimens in patients with newly diagnosed Ewing sarcoma (EE2012): an open-label, randomised, phase 3 trial. Lancet 400 (10362): 1513-1521, 2022. [PUBMED Abstract]
- Ferrari S, Mercuri M, Rosito P, et al.: Ifosfamide and actinomycin-D, added in the induction phase to vincristine, cyclophosphamide and doxorubicin, improve histologic response and prognosis in patients with non metastatic Ewing's sarcoma of the extremity. J Chemother 10 (6): 484-91, 1998. [PUBMED Abstract]
- Kolb EA, Kushner BH, Gorlick R, et al.: Long-term event-free survival after intensive chemotherapy for Ewing's family of tumors in children and young adults. J Clin Oncol 21 (18): 3423-30, 2003. [PUBMED Abstract]
- Granowetter L, Womer R, Devidas M, et al.: Dose-intensified compared with standard chemotherapy for nonmetastatic Ewing sarcoma family of tumors: a Children's Oncology Group Study. J Clin Oncol 27 (15): 2536-41, 2009. [PUBMED Abstract]
- Cash T, Krailo MD, Buxton AB, et al.: Long-Term Outcomes in Patients With Localized Ewing Sarcoma Treated With Interval-Compressed Chemotherapy on Children's Oncology Group Study AEWS0031. J Clin Oncol 41 (30): 4724-4728, 2023. [PUBMED Abstract]
- Koch R, Haveman L, Ladenstein R, et al.: Zoledronic Acid Add-on Therapy for Standard-Risk Ewing Sarcoma Patients in the Ewing 2008R1 Trial. Clin Cancer Res 29 (24): 5057-5068, 2023. [PUBMED Abstract]
- DuBois SG, Krailo MD, Gebhardt MC, et al.: Comparative evaluation of local control strategies in localized Ewing sarcoma of bone: a report from the Children's Oncology Group. Cancer 121 (3): 467-75, 2015. [PUBMED Abstract]
- Hoffmann C, Ahrens S, Dunst J, et al.: Pelvic Ewing sarcoma: a retrospective analysis of 241 cases. Cancer 85 (4): 869-77, 1999. [PUBMED Abstract]
- Shamberger RC, Laquaglia MP, Krailo MD, et al.: Ewing sarcoma of the rib: results of an intergroup study with analysis of outcome by timing of resection. J Thorac Cardiovasc Surg 119 (6): 1154-61, 2000. [PUBMED Abstract]
- Oberlin O, Deley MC, Bui BN, et al.: Prognostic factors in localized Ewing's tumours and peripheral neuroectodermal tumours: the third study of the French Society of Paediatric Oncology (EW88 study). Br J Cancer 85 (11): 1646-54, 2001. [PUBMED Abstract]
- Yock TI, Krailo M, Fryer CJ, et al.: Local control in pelvic Ewing sarcoma: analysis from INT-0091--a report from the Children's Oncology Group. J Clin Oncol 24 (24): 3838-43, 2006. [PUBMED Abstract]
- Schlegel M, Zeumer M, Prodinger PM, et al.: Impact of Pathological Fractures on the Prognosis of Primary Malignant Bone Sarcoma in Children and Adults: A Single-Center Retrospective Study of 205 Patients. Oncology 94 (6): 354-362, 2018. [PUBMED Abstract]
- Bramer JA, Abudu AA, Grimer RJ, et al.: Do pathological fractures influence survival and local recurrence rate in bony sarcomas? Eur J Cancer 43 (13): 1944-51, 2007. [PUBMED Abstract]
- Krasin MJ, Rodriguez-Galindo C, Billups CA, et al.: Definitive irradiation in multidisciplinary management of localized Ewing sarcoma family of tumors in pediatric patients: outcome and prognostic factors. Int J Radiat Oncol Biol Phys 60 (3): 830-8, 2004. [PUBMED Abstract]
- Gerrand C, Bate J, Seddon B, et al.: Seeking international consensus on approaches to primary tumour treatment in Ewing sarcoma. Clin Sarcoma Res 10 (1): 21, 2020. [PUBMED Abstract]
- Laskar S, Sinha S, Chatterjee A, et al.: Radiation Therapy Dose Escalation in Unresectable Ewing Sarcoma: Final Results of a Phase 3 Randomized Controlled Trial. Int J Radiat Oncol Biol Phys 113 (5): 996-1002, 2022. [PUBMED Abstract]
- Kacar M, Nagel MB, Liang J, et al.: Radiation therapy dose escalation achieves high rates of local control with tolerable toxicity profile in pediatric and young adult patients with Ewing sarcoma. Cancer 130 (10): 1836-1843, 2024. [PUBMED Abstract]
- Rombi B, DeLaney TF, MacDonald SM, et al.: Proton radiotherapy for pediatric Ewing's sarcoma: initial clinical outcomes. Int J Radiat Oncol Biol Phys 82 (3): 1142-8, 2012. [PUBMED Abstract]
- Fuchs B, Valenzuela RG, Sim FH: Pathologic fracture as a complication in the treatment of Ewing's sarcoma. Clin Orthop (415): 25-30, 2003. [PUBMED Abstract]
- Ahmed SK, Witten BG, Harmsen WS, et al.: Analysis of Local Control Outcomes and Clinical Prognostic Factors in Localized Pelvic Ewing Sarcoma Patients Treated With Radiation Therapy: A Report From the Children's Oncology Group. Int J Radiat Oncol Biol Phys 115 (2): 337-346, 2023. [PUBMED Abstract]
- Schuck A, Ahrens S, Konarzewska A, et al.: Hemithorax irradiation for Ewing tumors of the chest wall. Int J Radiat Oncol Biol Phys 54 (3): 830-8, 2002. [PUBMED Abstract]
- Kuttesch JF, Wexler LH, Marcus RB, et al.: Second malignancies after Ewing's sarcoma: radiation dose-dependency of secondary sarcomas. J Clin Oncol 14 (10): 2818-25, 1996. [PUBMED Abstract]
- Whelan J, Le Deley MC, Dirksen U, et al.: High-Dose Chemotherapy and Blood Autologous Stem-Cell Rescue Compared With Standard Chemotherapy in Localized High-Risk Ewing Sarcoma: Results of Euro-E.W.I.N.G.99 and Ewing-2008. J Clin Oncol : JCO2018782516, 2018. [PUBMED Abstract]
- Dirksen U, Brennan B, Le Deley MC, et al.: High-Dose Chemotherapy Compared With Standard Chemotherapy and Lung Radiation in Ewing Sarcoma With Pulmonary Metastases: Results of the European Ewing Tumour Working Initiative of National Groups, 99 Trial and EWING 2008. J Clin Oncol 37 (34): 3192-3202, 2019. [PUBMED Abstract]
- Le Deley MC, Paulussen M, Lewis I, et al.: Cyclophosphamide compared with ifosfamide in consolidation treatment of standard-risk Ewing sarcoma: results of the randomized noninferiority Euro-EWING99-R1 trial. J Clin Oncol 32 (23): 2440-8, 2014. [PUBMED Abstract]
Tratamiento del sarcoma de Ewing metastásico
Alrededor del 25 % de los pacientes con sarcoma de Ewing tienen metástasis en el momento del diagnóstico.[1] El pronóstico es precario para los pacientes con enfermedad metastásica. Con los tratamientos actuales, los pacientes con enfermedad metastásica tienen una tasa de supervivencia sin complicaciones (SSC) a 6 años de alrededor del 28 % y una tasa de supervivencia general (SG) de cerca del 30 %.[2,3] Para los pacientes con metástasis pulmonares o pleurales solas, la tasa de SSC a 6 años es de alrededor del 40 % cuando se usa irradiación pulmonar bilateral.[2,4] En contraste, los pacientes con metástasis óseas o en la médula ósea tienen una tasa de SSC a 4 años de alrededor del 28 %, y los pacientes con metástasis combinadas en los pulmones y los huesos y la médula ósea tienen una tasa de SSC a 4 años de alrededor del 14 %.[4,5]
Los siguientes factores predicen de manera independiente un desenlace precario en pacientes que presentan enfermedad metastásica:[3]
- Edad mayor de 14 años.
- Volumen del tumor primario de más de 200 ml.
- Más de un sitio de metástasis óseas.
- Metástasis en la médula ósea.
- Varias metástasis pulmonares.
- Las metástasis pulmonares carecen de respuesta completa a la quimioterapia de inducción.[6]
Las opciones de tratamiento estándar del sarcoma de Ewing metastásico son las siguientes:
Quimioterapia
En los pacientes con sarcoma de Ewing metastásico, el tratamiento estándar en el que se alternan ciclos de vincristina, doxorrubicina y ciclofosfamida con ifosfamida y etopósido (VDC/IE) combinados con medidas de control local adecuadas en el sitio primario y metastásico, suele producir respuestas completas o parciales. Sin embargo, la tasa curación general es del 20 %.[5,7,8]
Los siguientes regímenes de quimioterapia no han demostrado beneficios:
- En el Intergroup Ewing Sarcoma Study, los pacientes con enfermedad metastásica no mostraron ningún beneficio con la adición de ifosfamida y etopósido a un régimen estándar de vincristina, doxorrubicina, ciclofosfamida y dactinomicina.[8]
- En otro análisis de este estudio, el aumento en la intensidad de la dosis de ciclofosfamida, ifosfamida y doxorrubicina no mejoró el desenlace, en comparación con los regímenes con dosis de intensidad estándar. Este régimen aumentó la toxicidad y el riesgo de una segunda neoplasia maligna sin mejorar la SSC ni la SG.[2]
- La intensificación de ifosfamida a 2,8 g/m2 diarios durante 5 días no mejoró el desenlace cuando se administró con quimioterapia estándar a pacientes con sarcoma de Ewing metastásico recién diagnosticado.[9][Nivel de evidencia C2]
- En un estudio piloto de dosis bajas de terapia antiangiogénica con vinblastina y celecoxib, que se añadió a la terapia de 3 semanas con VDC/IE, no se mejoraron los desenlaces de los pacientes que presentaron metástasis.[10]
- El Children's Oncology Group realizó un ensayo aleatorizado prospectivo (AEWS1221 [NCT02306161]) en el que se probó la adición de ganitumab a la quimioterapia multifarmacológica. En el momento de la inscripción, los pacientes se asignaron al azar (1:1) al grupo estándar (VDC/IE de intervalos reducidos) o al grupo experimental (se administra ganitumab con VDC/IE cuando comienza el ciclo y en monoterapia una vez cada 3 semanas por 6 meses después del tratamiento convencional). En el estudio se incluyeron 298 pacientes aptos (148 en el grupo estándar; 150 en el grupo experimental).[11][Nivel de evidencia B1]
- Las estimaciones de la tasa de SSC a 3 años fueron del 37,4 % (intervalo de confianza [IC] 95 %, 29,3–45,5 %) en los pacientes del grupo estándar y del 39,1 % (IC 95 %, 31,3–46,7 %) en los pacientes del grupo experimental (cociente de riesgos instantáneos [CRI] estratificado para un evento de SSC en el grupo experimental, 1,00; IC 95 %, 0,76–1,33; unilateral, P = 0,50).
- Los pacientes del grupo experimental notificaron más casos de neumonitis posterior a la radioterapia con exposición de campos torácicos y tasas nominalmente más altas de neutropenia febril y elevación de la alanina–transaminasa.
- La incorporación de ganitumab a la quimioterapia de intervalos reducidos no redujo de manera significativa el riesgo de un evento de SSC en los pacientes con sarcoma de Ewing metastásico recién diagnosticado. Los desenlaces de estos pacientes fueron similares a los de ensayos anteriores que no recibieron inhibición de IGF-1R ni compresión por intervalos.
- La incorporación de ganitumab quizás se relacione con un aumento de los efectos tóxicos.
Cirugía y radioterapia
El uso sistemático de radioterapia y cirugía para los sitios de metástasis puede mejorar el resultado general en pacientes con metástasis extrapulmonares, aunque no se ha realizado ningún ensayo aleatorizado.
Evidencia (cirugía y radioterapia):
- En un análisis retrospectivo de datos de 120 pacientes con sarcoma de Ewing metastásico multifocal, los pacientes que recibieron tratamiento local para el tumor primario y las metástasis presentaron mejores desenlaces que quienes recibieron tratamiento local solo en el tumor primario o que no recibieron tratamiento local (tasa de SSC a 3 años, 39 % vs. 17 % y 14 %, respectivamente; P < 0,001).[12]
- En 3 análisis retrospectivos de grupos más pequeños de pacientes que recibieron radioterapia dirigida a todos los sitios tumorales, se observó una tendencia similar con mejores desenlaces con la irradiación de todos los sitios de enfermedad metastásica.[13-15]
Estos resultados se deben interpretar con prudencia. Los pacientes que recibieron terapia de control local en todos los sitios conocidos con enfermedad metastásica fueron seleccionados por el investigador que administraba el tratamiento, no se asignaron al azar. Los pacientes con tantas metástasis que la radioterapia a todos los sitios produciría una insuficiencia de la médula ósea no se asignaron a recibir radiación a todos los sitios de enfermedad metastásica. Los pacientes que no alcanzaron el control del tumor primario no continuaron recibiendo terapia de control local en todos los sitios de enfermedad metastásica. Hubo un sesgo de selección. Aunque todos los pacientes en estos informes presentaban múltiples sitios de enfermedad metastásica, aquellos que se sometieron a cirugía o radioterapia dirigida a todos los sitios de enfermedad metastásica clínicamente detectables presentaron mejores respuestas al tratamiento sistémico y menos sitios de metástasis que los que no recibieron un tratamiento similar en los sitios metastásicos.
Se deberá considerar la administración de radioterapia en un centro en el que expertos en el tratamiento del sarcoma de Ewing apliquen técnicas estrictas de planificación. Este abordaje dará lugar a control local del tumor con morbilidad aceptable en la mayoría de los pacientes.[16]
La dosis de radiación depende del sitio de la enfermedad metastásica:
- Huesos y tejido blandos. Se ha utilizado la radioterapia corporal estereotáctica para tratar sitios metastásicos en huesos y tejidos blandos. La mediana de dosis total de radioterapia corporal estereotáctica curativa o definitiva administrada fue de 40 Gy en 5 fracciones (intervalo, 30–60 Gy en 3–10 fracciones). La mediana de dosis total de radioterapia corporal estereotáctica paliativa administrada fue de 40 Gy en 5 fracciones (intervalo, 16–50 Gy en 1–10 fracciones). Estos regímenes de cursos cortos con fracciones de dosis grandes son el equivalente biológico a dosis más altas administradas en fracciones de dosis más pequeñas durante cursos de tratamiento más prolongados.[17][Nivel de evidencia C1]
- Pulmones. Para todos los pacientes con metástasis pulmonares, se debe considerar la irradiación dirigida a todo el pulmón, incluso si se logró la resolución completa de la enfermedad pulmonar metastásica manifiesta con quimioterapia.[4,5,18] Las dosis de radiación se modulan de acuerdo con el tamaño del pulmón que se irradiará y con el funcionamiento pulmonar. Por lo general, se usan dosis de 12 Gy a 15 Gy cuando se tratan los pulmones completos. No se ha realizado ningún ensayo aleatorizado para demostrar que la radioterapia mejora la supervivencia en este grupo de pacientes. En un ensayo preliminar del grupo intergrupal de sarcoma de Ewing en la década de 1970, se observó que la radiación dirigida al pulmón mejoró la supervivencia en pacientes con enfermedad no metastásica cuando se agregó vincristina, actinomicina y ciclofosfamida (VAC), en comparación con VAC solo.[19] Sin embargo, la adición de doxorrubicina al tratamiento produjo una SSC mejor que el régimen de VAC con radioterapia.
Otros tratamientos
Los tratamientos más intensivos, muchos de los cuales incorporan dosis altas de quimioterapia con irradiación total del cuerpo o sin esta con apoyo de células madre, no lograron mejoras en las tasas de SSC de pacientes con metástasis óseas o en la médula ósea.[2,3,13,20-22]; [23][Nivel de evidencia C2] Para obtener más información, consultar la sección Quimioterapia de dosis altas con apoyo de células madre para el sarcoma de Ewing.
-
Quimioterapia de dosis altas con apoyo de células madre.
- El EWING 2008R3 (NCT00987636) fue el primer ensayo controlado multicéntrico, sin enmascaramiento, aleatorizado y de fase III dirigido a pacientes recién diagnosticados con sarcoma de Ewing diseminado que presentaban metástasis óseas o en otros sitios.[24][Nivel de evidencia B1] En el estudio se evaluó el efecto en la SSC y SG del tratamiento con quimioterapia de dosis altas de treosulfano y melfalán (TreoMel-HDT) seguida de infusión de células madre hematopoyéticas autógenas. Después de 6 ciclos de quimioterapia (vincristina, ifosfamida, doxorrubicina y etopósido), los pacientes que otorgaron el consentimiento se asignaron al azar a recibir 8 ciclos de terapia de consolidación (vincristina, dactinomicina y ciclofosfamida) o terapia de consolidación con TreoMel-HDT seguida por reinfusión de células madre autógenas. Entre 2009 y 2018, 109 pacientes se asignaron al azar, 55 a recibir TreoMel-HDT. No se encontraron diferencias significativas en las tasas de SSC a 3 años entre los pacientes que recibieron TreoMel-HDT y los pacientes del grupo de control (20,9 vs. 19,2 %; mediana de seguimiento, 3,3 años).
- Uno de los estudios más grandes fue el ensayo EURO-EWING-INTERGROUP-EE99 R3 en el que participaron 281 pacientes con sarcoma de Ewing primario con diseminación metastásica. Los pacientes se trataron con 6 ciclos de vincristina, ifosfamida, doxorrubicina y etopósido seguidos de terapia de dosis altas y trasplante autógeno de células madre. Los pacientes tuvieron una tasa de SSC a 3 años del 27 % y una tasa de SG del 34 %. Los factores pronósticos independientes identificados fueron la presencia y el número de lesiones óseas, un volumen del tumor primario mayor de 200 ml, edad mayor de 14 años, múltiples metástasis pulmonares y compromiso de la médula ósea.[3][Nivel de evidencia C2]
- La función de la terapia con dosis altas de busulfano y melfalán (BuMel) seguida de rescate de células madre se investigó en el ensayo prospectivo, aleatorizado EURO-EWING-INTERGROUP-EE99 (NCT00020566). Entre los pacientes con metástasis pulmonares aisladas, no hubo ninguna diferencia en las tasas de SSC a 3 años (55,7 % con BuMel vs. 50,3 % con quimioterapia continuada y radioterapia dirigida a todo el pulmón; P = 0,21).[25]
- Melfalán. El melfalán, en dosis que no causen mielosupresión, demostró ser un fármaco activo en un estudio de tratamiento durante el intervalo inicial para pacientes con enfermedad metastásica en el momento del diagnóstico; sin embargo, la tasa de curación fue muy baja.[26]
- Irinotecán. Se administró irinotecán en monoterapia en un estudio de tratamiento durante el intervalo inicial para pacientes con sarcoma de Ewing metastásico recién diagnosticado y se observó una eficacia relativamente baja (respuesta parcial en 5 de 24 pacientes).[27][Nivel de evidencia C3] Es necesario investigar más para determinar la dosificación del irinotecán y sus combinaciones con otros fármacos en los pacientes con sarcoma de Ewing.
Ensayos clínicos en curso
Realizar una búsqueda avanzada en inglés de los ensayos clínicos sobre cáncer auspiciados por el NCI que ahora aceptan pacientes. La búsqueda se puede simplificar por ubicación del ensayo, tipo de tratamiento, nombre del fármaco y otros criterios. También se dispone de información general sobre los ensayos clínicos.
Bibliografía
- Esiashvili N, Goodman M, Marcus RB: Changes in incidence and survival of Ewing sarcoma patients over the past 3 decades: Surveillance Epidemiology and End Results data. J Pediatr Hematol Oncol 30 (6): 425-30, 2008. [PUBMED Abstract]
- Miser JS, Goldsby RE, Chen Z, et al.: Treatment of metastatic Ewing sarcoma/primitive neuroectodermal tumor of bone: evaluation of increasing the dose intensity of chemotherapy--a report from the Children's Oncology Group. Pediatr Blood Cancer 49 (7): 894-900, 2007. [PUBMED Abstract]
- Ladenstein R, Pötschger U, Le Deley MC, et al.: Primary disseminated multifocal Ewing sarcoma: results of the Euro-EWING 99 trial. J Clin Oncol 28 (20): 3284-91, 2010. [PUBMED Abstract]
- Paulussen M, Ahrens S, Craft AW, et al.: Ewing's tumors with primary lung metastases: survival analysis of 114 (European Intergroup) Cooperative Ewing's Sarcoma Studies patients. J Clin Oncol 16 (9): 3044-52, 1998. [PUBMED Abstract]
- Paulussen M, Ahrens S, Burdach S, et al.: Primary metastatic (stage IV) Ewing tumor: survival analysis of 171 patients from the EICESS studies. European Intergroup Cooperative Ewing Sarcoma Studies. Ann Oncol 9 (3): 275-81, 1998. [PUBMED Abstract]
- Halalsheh H, Kaste SC, Krasin MJ, et al.: Clinical impact of post-induction resolution of pulmonary lesions in metastatic Ewing sarcoma. Pediatr Blood Cancer 67 (4): e28150, 2020. [PUBMED Abstract]
- Pinkerton CR, Bataillard A, Guillo S, et al.: Treatment strategies for metastatic Ewing's sarcoma. Eur J Cancer 37 (11): 1338-44, 2001. [PUBMED Abstract]
- Miser JS, Krailo MD, Tarbell NJ, et al.: Treatment of metastatic Ewing's sarcoma or primitive neuroectodermal tumor of bone: evaluation of combination ifosfamide and etoposide--a Children's Cancer Group and Pediatric Oncology Group study. J Clin Oncol 22 (14): 2873-6, 2004. [PUBMED Abstract]
- Magnan H, Goodbody CM, Riedel E, et al.: Ifosfamide dose-intensification for patients with metastatic Ewing sarcoma. Pediatr Blood Cancer 62 (4): 594-7, 2015. [PUBMED Abstract]
- Felgenhauer JL, Nieder ML, Krailo MD, et al.: A pilot study of low-dose anti-angiogenic chemotherapy in combination with standard multiagent chemotherapy for patients with newly diagnosed metastatic Ewing sarcoma family of tumors: A Children's Oncology Group (COG) Phase II study NCT00061893. Pediatr Blood Cancer 60 (3): 409-14, 2013. [PUBMED Abstract]
- DuBois SG, Krailo MD, Glade-Bender J, et al.: Randomized Phase III Trial of Ganitumab With Interval-Compressed Chemotherapy for Patients With Newly Diagnosed Metastatic Ewing Sarcoma: A Report From the Children's Oncology Group. J Clin Oncol 41 (11): 2098-2107, 2023. [PUBMED Abstract]
- Haeusler J, Ranft A, Boelling T, et al.: The value of local treatment in patients with primary, disseminated, multifocal Ewing sarcoma (PDMES). Cancer 116 (2): 443-50, 2010. [PUBMED Abstract]
- Burdach S, Thiel U, Schöniger M, et al.: Total body MRI-governed involved compartment irradiation combined with high-dose chemotherapy and stem cell rescue improves long-term survival in Ewing tumor patients with multiple primary bone metastases. Bone Marrow Transplant 45 (3): 483-9, 2010. [PUBMED Abstract]
- Paulino AC, Mai WY, Teh BS: Radiotherapy in metastatic ewing sarcoma. Am J Clin Oncol 36 (3): 283-6, 2013. [PUBMED Abstract]
- Casey DL, Wexler LH, Meyers PA, et al.: Radiation for bone metastases in Ewing sarcoma and rhabdomyosarcoma. Pediatr Blood Cancer 62 (3): 445-9, 2015. [PUBMED Abstract]
- Donaldson SS, Torrey M, Link MP, et al.: A multidisciplinary study investigating radiotherapy in Ewing's sarcoma: end results of POG #8346. Pediatric Oncology Group. Int J Radiat Oncol Biol Phys 42 (1): 125-35, 1998. [PUBMED Abstract]
- Brown LC, Lester RA, Grams MP, et al.: Stereotactic body radiotherapy for metastatic and recurrent ewing sarcoma and osteosarcoma. Sarcoma 2014: 418270, 2014. [PUBMED Abstract]
- Spunt SL, McCarville MB, Kun LE, et al.: Selective use of whole-lung irradiation for patients with Ewing sarcoma family tumors and pulmonary metastases at the time of diagnosis. J Pediatr Hematol Oncol 23 (2): 93-8, 2001. [PUBMED Abstract]
- Nesbit ME, Perez CA, Tefft M, et al.: Multimodal therapy for the management of primary, nonmetastatic Ewing's sarcoma of bone: an Intergroup Study. Natl Cancer Inst Monogr (56): 255-62, 1981. [PUBMED Abstract]
- Meyers PA, Krailo MD, Ladanyi M, et al.: High-dose melphalan, etoposide, total-body irradiation, and autologous stem-cell reconstitution as consolidation therapy for high-risk Ewing's sarcoma does not improve prognosis. J Clin Oncol 19 (11): 2812-20, 2001. [PUBMED Abstract]
- Burdach S, Meyer-Bahlburg A, Laws HJ, et al.: High-dose therapy for patients with primary multifocal and early relapsed Ewing's tumors: results of two consecutive regimens assessing the role of total-body irradiation. J Clin Oncol 21 (16): 3072-8, 2003. [PUBMED Abstract]
- Thiel U, Wawer A, Wolf P, et al.: No improvement of survival with reduced- versus high-intensity conditioning for allogeneic stem cell transplants in Ewing tumor patients. Ann Oncol 22 (7): 1614-21, 2011. [PUBMED Abstract]
- Loschi S, Dufour C, Oberlin O, et al.: Tandem high-dose chemotherapy strategy as first-line treatment of primary disseminated multifocal Ewing sarcomas in children, adolescents and young adults. Bone Marrow Transplant 50 (8): 1083-8, 2015. [PUBMED Abstract]
- Koch R, Gelderblom H, Haveman L, et al.: High-Dose Treosulfan and Melphalan as Consolidation Therapy Versus Standard Therapy for High-Risk (Metastatic) Ewing Sarcoma. J Clin Oncol 40 (21): 2307-2320, 2022. [PUBMED Abstract]
- Whelan J, Le Deley MC, Dirksen U, et al.: High-Dose Chemotherapy and Blood Autologous Stem-Cell Rescue Compared With Standard Chemotherapy in Localized High-Risk Ewing Sarcoma: Results of Euro-E.W.I.N.G.99 and Ewing-2008. J Clin Oncol : JCO2018782516, 2018. [PUBMED Abstract]
- Luksch R, Grignani G, Fagioli F, et al.: Response to melphalan in up-front investigational window therapy for patients with metastatic Ewing's family tumours. Eur J Cancer 43 (5): 885-90, 2007. [PUBMED Abstract]
- Morland B, Platt K, Whelan JS: A phase II window study of irinotecan (CPT-11) in high risk Ewing sarcoma: a Euro-E.W.I.N.G. study. Pediatr Blood Cancer 61 (3): 442-5, 2014. [PUBMED Abstract]
Tratamiento del sarcoma de Ewing recidivante
La recidiva del sarcoma de Ewing es más común dentro de los 2 años posteriores al diagnóstico inicial (cerca del 80 %).[1,2] Sin embargo, se presentan recaídas tardías después de 5 años del diagnóstico inicial, que son más comunes en el sarcoma de Ewing (13 %; intervalo de confianza 95 %, 9,4–16,5 %) que en otros tumores sólidos infantiles.[3] En un análisis de la base de datos del Programa de Vigilancia, Epidemiología y Resultados Finales (SEER) se identificaron 1351 pacientes que sobrevivieron más de 60 meses desde el diagnóstico.[4] De estos pacientes, 209 murieron; 144 de las muertes (69 %) se atribuyeron a sarcoma de Ewing progresivo recidivante. El riesgo más alto de muerte tardía se vinculó con la raza negra, el sexo masculino, una edad mayor en el momento del diagnóstico inicial, y tumores primarios óseos de la pelvis y el esqueleto axial Este análisis abarcó el período entre 1973 y 2013, y los 1351 pacientes solo representaban el 38 % de los pacientes de la muestra original; esto refleja los desenlaces más precarios de los tratamientos en épocas anteriores. Es posible que los pacientes que alcanzan el punto de supervivencia de 5 años tras recibir el tratamiento más contemporáneo no repitan esta experiencia.
El pronóstico general de los pacientes con sarcoma de Ewing recidivante es precario. La tasa de supervivencia a 5 años después de la recidiva es de alrededor del 10 % al 15 %.[2,5,6]; [1][Nivel de evidencia C1] Los pacientes con sarcoma de Ewing en recaída o progresivo con enfermedad cuantificable tienen una tasa de supervivencia sin complicaciones (SSC) a 6 meses del 13 %.[7][Nivel de evidencia C1]
Los siguientes son los factores pronósticos:
- Tiempo hasta la recidiva. El tiempo hasta la recidiva es el factor pronóstico más importante. Los pacientes cuyo sarcoma de Ewing recidivó más de 2 años después del diagnóstico inicial tuvieron una tasa de supervivencia a 5 años de un 30 %, en comparación con un 7 % en los pacientes cuya enfermedad recidivó en el transcurso de 2 años.[1,2]
- Recidiva local y a distancia. Los pacientes que presentan recidiva local y metástasis a distancia tienen desenlaces más precarios que aquellos con recidiva local aislada o recidiva metastásica sola.[1,2]
- Recidiva pulmonar aislada. La recidiva pulmonar aislada no fue un factor pronóstico importante en una serie de América del Norte.[1] En las experiencias italiana o escandinava, la edad más temprana, el intervalo sin enfermedad más prolongado y la recidiva solo en el pulmón se relacionaron con una supervivencia sin progresión (SSP) más prolongada después de la recidiva. En esta experiencia, fue menos probable que los pacientes con una recidiva de sarcoma de Ewing después del tratamiento inicial con dosis altas y rescate autógeno de células madre lograran una segunda remisión completa.[8][Nivel de evidencia C2]
La selección del tratamiento para pacientes con enfermedad recidivante depende de muchos factores como los siguientes:
- Sitio de la recidiva.
- Tratamiento previo.
- Consideraciones individuales del paciente.
No hay un tratamiento de segunda línea estándar para todos los pacientes con sarcoma de Ewing en recaída o resistente al tratamiento. En el momento de la primera recaída, la mayoría de los pacientes se tratan con quimioterapia sistémica convencional. Los pacientes que muestran una respuesta al tratamiento a veces se someten al control local de los sitios de recidiva.
Las opciones de tratamiento estándar para el sarcoma de Ewing recidivante son las siguientes:
Quimioterapia
Las combinaciones de quimioterapia, como ciclofosfamida y topotecán, o irinotecán y temozolomida, con vincristina o sin esta, son eficaces para el sarcoma de Ewing recidivante y son opciones a considerar para estos pacientes.[9-14]
| Referencia del estudio | Fase del ensayo (Número total de pacientes) | Mediana de edad (Intervalo) (años) | RC/RP | RR | Ciclofosfamida (mg/m2)/topotecán (mg/m2) x día | Otros fármacos |
|---|---|---|---|---|---|---|
| II = ensayo de fase II; RC = respuesta completa; RP = respuesta parcial; R = retrospectivo; RR = tasa de respuesta objetiva; VCR = vincristina. | ||||||
| Saylors et al.[9] | II (17) | 13,8 (1–21) | 1/3 | 29 % | 250 × 5/0,75 × 5 | Ninguno |
| Hunold et al.[11] | R (54) | 17,4 (3–49) | 0/16 | 30 % | 250 × 5/0,75 × 5 | Ninguno |
| Farhat et al.[15] | R (14) | 11 (2–19) | 0/3 | 21 % | 250 × 5/0,75 × 5 | Ninguno |
| Kebudi et al.[16] | R (14) | 13 (3–16) | 2/5 | 50 % | 250 × 5/0,75 × 5 | VCR |
Estos estudios fueron retrospectivos. Faltan ensayos prospectivos con cohortes de elegibilidad claramente definidas y análisis por intención de tratar. En combinación, en estos estudios participaron 99 pacientes y se observaron 3 remisiones completas y 27 remisiones parciales. La tasa de respuesta objetiva fue del 30 %.
| Referencia del estudio | Fase del ensayo (Número total de pacientes) | Mediana de edad (Intervalo) (años) | RC/RP | RR | Temozolomida (mg/m2)/irinotecán (mg/m2) x día x semana | Otros fármacos |
|---|---|---|---|---|---|---|
| I = ensayo de fase I; II = ensayo de fase II; Acta Onc = Acta Oncológica; An Ped = Annals of Pediatrics; Clin Cancer Res = Clinical Cancer Research; Exp Opin = Expert Opinion Investigational Drugs; Clin Transl Oncol = Clinical and Translational Oncology; BEV = bevacizumab; IV = intravenoso; N/A = no aplicable; PBC = Pediatric Blood and Cancer; Ped Hem Onc = Pediatric Hematology and Oncology; VO = vía oral; R = ensayo retrospectivo; RR = tasa de respuesta objetiva; TMS = temsirólimus; VCR = vincristina. | ||||||
| Wagner et al. (PBC, 2007) [12] | R (16) | 18 (7–33) | 1/3 | 29 % | 100 × 5/IV 10–20 × 5 × 2 | Ninguno |
| Casey et al. (PBC, 2009) [13] | R (19) | 19,5 (2–40) | 5/7 | 63 % | 100 × 5/IV 20 × 5 × 2 | Ninguno |
| Hernandez-Marques et al. (An Ped, 2013) [17] | R (8) | 13 (6–18) | 0/3 | 37 % | 80–100 × 5/IV 10–20 × 5 × 2 | Ninguno |
| Raciborska et al. (PBC, 2013) [14] | R (22) | 14,3 | 5/7 | 54 % | 125 × 5/IV 50 × 5 | VCR |
| McKnall-Knapp et al. (PBC, 2010) [18] | I (1) | N/A | 0/1 | 100 % | 100 × 5/IV 20 × 5 × 2 | VCR |
| Wagner et al. (PBC, 2010) [19] | I (5) | (<21) | 1/1 | 40 % | 100–150 × 5/VO 35–90 × 5 | VCR |
| Wagner et al. (PBC, 2013) [20] | I (2) | 20, 22 | 1/1 | 100 % | 150 × 5/VO 90 × 5 | VCR, BEV |
| Bagatell et al. (PBC, 2014) [21] | 7 | (<21) | 0/1 | 14 % | 100–150 × 5/VO 50–90 × 5 | TMS |
| Kurucu et al. (Ped Hem Onc, 2015) [22] | R (20) | 14 (1–18) | Se desconoce | 55 % | 100 × 5/IV 20 × 5 × 2 | Ninguno |
| Anderson et al. (Exp Opin, 2008) [23] | R (25) | 15 | 7/9 | 64 % | 100 × 5/IV 10 × 5 × 2 | Ninguno |
| Palmerini et al. (Acta Onc, 2018) [24] | R (51) | 21 (3–65) | 5/12 | 34 % | 100 × 5/IV 40 × 5 | Ninguno |
| Salah et al. (Clin Transl Oncol, 2021) [25] | R (53) | 20 (5–45) | 1/11 | 28 % | 100 × 5/IV 40 × 5 en 21 pacientes; IV 50 × 5 en 24 pacientes; IV 20 × 5 × 2 en 6 pacientes | Ninguno |
| Xu et al. (Clin Cancer Res, 2023) [26] programa de 5 días: | II (24) | 16,.5 ± 7,9 | 1/4 | 20,8 % | 100 × 5/50 × 5 | VCR 1,4 mg/m2 día 1 |
| Xu et al. (Clin Cancer Res, 2023) [26] programa de 10 días: | II (22) | 15,2 ± 6,3 | 1/11 | 54,5 % | 100 × 5/20 × 5 × 2 | VCR 1,4 mg/m2 días 1 y 8 |
La mayoría de estos estudios fueron retrospectivos, no prospectivos. Solo hay 4 ensayos prospectivos con cohortes de elegibilidad bien definidas y con informe por intención de tratar. Además, hay mucha variabilidad en los informes sobre las dosis y regímenes de dosificación de irinotecán y temozolomida, así como sobre el uso de otros fármacos. En combinación, en estos estudios participaron 275 pacientes y se observaron 21 remisiones completas y 82 remisiones parciales. La tasa de respuesta objetiva fue del 37,5 %.
Evidencia (quimioterapia):
- En un estudio de fase II de topotecán y ciclofosfamida, se observó una respuesta en 6 de 17 pacientes con sarcoma de Ewing.[9] En un ensayo similar en Alemania, 16 de 49 pacientes presentaron una respuesta clínica.[11]
- En varios estudios retrospectivos se demostró la actividad de la temozolomida y el irinotecán en pacientes con sarcoma de Ewing recidivante.[13,22,24]
- En el mayor estudio retrospectivo multicéntrico sobre la combinación de temozolomida e irinotecán en pacientes con sarcoma de Ewing recidivante y resistente al tratamiento primario, 51 pacientes (66 % de los pacientes ≥18 años; mediana de edad, 21 años) recibieron temozolomida (100 mg/m2/día oral) e irinotecán (40 mg/2/día intravenoso), los días 1 a 5, cada 21 días. El 25 % de los pacientes presentaban una primera recaída o progresión, mientras que el resto de los pacientes presentaban una segunda recaída o una recaída superior.[24]
- Se observaron remisiones completas en 5 pacientes (10 %), remisiones parciales en 12 pacientes (24 %) y enfermedad estable en 19 pacientes (37 %), con una tasa de control de la enfermedad del 71 %.
- En el análisis univariante, los únicos 2 factores que predijeron la respuesta a temozolomida e irinotecán en cuanto a la SSP fueron el índice de desempeño y las concentraciones de lactato–deshidrogenasa.
- Se volvió a administrar temozolomida e irinotecán a 2 pacientes después de que se logró inducir la remisión de la enfermedad. Ambos pacientes lograron remisiones parciales en la reexposición. La remisión de un paciente se prolongó, por lo menos, 15 ciclos y la remisión del otro 22 ciclos.[24]
- En un ensayo prospectivo aleatorizado se compararon 2 programas de irinotecán administrados, en combinación con vincristina y temozolomida, para el tratamiento del sarcoma de Ewing recidivante. En una de las pautas se administraron 50 mg/m2 de irinotecán diarios durante 5 días (d × 5) y en la otra pauta se administraron 20 mg/m2 de irinotecán diarios por 5 días por 2 semanas consecutivas (10 dosis; d x 5 x 2).[26]
- La tasa de respuesta objetiva a las 12 semanas fue inferior en los pacientes que se trataron con la pauta d × 5 (5 de 24; 20,8 %) que en los pacientes que se trataron con la pauta d × 5 × 2 (12 de 22; 54,5 %; P = 0,019).
- En una revisión retrospectiva de series publicadas se compararon los resultados del tratamiento con irinotecán y temozolomida en una pauta de 10 días con el tratamiento en una pauta de 5 días.[27]
- Entre los 89 pacientes tratados con la pauta de 10 días de irinotecán, hubo 47 respuestas objetivas (53 %).
- Entre 180 pacientes tratados con la pauta de 5 días de irinotecán, hubo 52 respuestas (29 %).
- En los 2 estudios en los que se usó la pauta de 5 días de irinotecán se notificaron medianas de tiempo hasta la progresión de 3,0 y 3,9 meses, respectivamente.
- En los 4 estudios en los que se usó la pauta de 10 días de irinotecán se notificaron medianas de tiempo hasta la progresión de 4,6, 5,5, 8,3, y 9,5 meses, respectivamente.
- En el mayor estudio retrospectivo multicéntrico sobre la combinación de temozolomida e irinotecán en pacientes con sarcoma de Ewing recidivante y resistente al tratamiento primario, 51 pacientes (66 % de los pacientes ≥18 años; mediana de edad, 21 años) recibieron temozolomida (100 mg/m2/día oral) e irinotecán (40 mg/2/día intravenoso), los días 1 a 5, cada 21 días. El 25 % de los pacientes presentaban una primera recaída o progresión, mientras que el resto de los pacientes presentaban una segunda recaída o una recaída superior.[24]
- Se han logrado respuestas objetivas en pacientes con sarcoma de Ewing en recaída con la combinación de docetaxel con gemcitabina o irinotecán.[28][Nivel de evidencia C1]; [29,30][Nivel de evidencia C3]
- Las dosis altas de ifosfamida (3 g/m2 por día durante 5 días = 15 g/m2) mostraron actividad en pacientes cuyo sarcoma de Ewing recidivó después del tratamiento que incluyó ifosfamida estándar (1,8 g/m2 por día durante 5 días = 9 g/m2).[31][Nivel de evidencia C3]
- Investigadores europeos están realizando un estudio prospectivo para comparar 4 regímenes para el tratamiento de pacientes con sarcoma de Ewing recidivante.[32] El ensayo multigrupo adaptativo de fase II/III es el primero en comparar regímenes en un diseño aleatorizado. Los pacientes de 4 a 50 años con sarcoma de Ewing resistente al tratamiento o recidivante, que estaban bastante sanos como para recibir quimioterapia, se asignaron al azar a recibir uno de 4 regímenes: topotecán y ciclofosfamida (TC), irinotecán y temozolomida (IT), gemcitabina y docetaxel (GD) o dosis altas de ifosfamida.[33]
- En una comparación pareada, se observó que la GD fue la menos eficaz.
- La respuesta en las pruebas por imágenes y los desenlaces de supervivencia después del uso de TC fueron ligeramente mejores que después de IT. Sin embargo, en una comparación de fase II, con la ifosfamida se obtuvo una SSP y una SG significativamente mejores que con la combinación TC. Como resultado, la ifosfamida fue el régimen más eficaz en este entorno, aunque con efectos tóxicos renales y neurológicos significativos.
- El estudio está en curso y se han inscrito más de 570 pacientes. En la actualidad, en el estudio se compara la ifosfamida y el lenvatinib con ifosfamida, carboplatino y etopósido.[34]
Tratamiento local para la recaída de la enfermedad
Cirugía
Es posible considerar la cirugía radical (amputación o hemipelvectomía) para los pacientes con sarcoma de Ewing local recidivante sin metástasis, aunque el pronóstico sea limitado.[35]
La función de la metastasectomía pulmonar en pacientes con enfermedad en recaída y metástasis pulmonares aisladas es polémica.[36,37]
Radioterapia
Se puede usar radioterapia (similar a las estrategias de primera línea) para los pacientes que recayeron después del inicio de la terapia de primera línea o que solo presentan metástasis pulmonares en recaída.[36]; [38][Nivel de evidencia C1] La radioterapia dirigida a lesiones óseas puede producir paliación, aunque la resección radical puede mejorar el desenlace.[2] Los pacientes con metástasis pulmonares que no recibieron radioterapia dirigida a los pulmones se deben considerar para recibir irradiación de todo el pulmón o radioterapia corporal estereotáctica.[36,39]; [38][Nivel de evidencia C1] En ocasiones, es posible extirpar quirúrgicamente la enfermedad residual pulmonar.
La paliación de las lesiones dolorosas en niños con enfermedad recidivante o progresiva se puede lograr con un curso corto (10 fracciones o menos) de radioterapia. En un estudio retrospectivo de 213 niños con diversas neoplasias malignas, que se trataron con estos cursos cortos de radioterapia, el 85 % de los pacientes experimentaron alivio del dolor completo o parcial, con pocos efectos tóxicos.[40]
Quimioterapia de dosis alta con apoyo de células madre
Aunque se han llevado a cabo intentos radicales para el control de la enfermedad, incluso regímenes mielosupresores, no hay evidencia que permita concluir que la terapia mielosupresora es superior a la quimioterapia estándar.[41-43]; [44][Nivel de evidencia C2]
La mayoría de los informes publicados sobre el uso de terapia de dosis altas y apoyo de células madre en pacientes con sarcoma de Ewing de riesgo alto tienen defectos metodológicos importantes. El error más común es la comparación de este grupo de riesgo alto con un grupo de control inapropiado. Los pacientes con sarcoma de Ewing que presentan un riesgo alto de fracaso del tratamiento y que recibieron terapia de dosis altas se comparan con pacientes que no recibieron terapia de dosis altas. Los pacientes que reciben terapia de dosis altas deben responder al tratamiento sistémico, permanecer vivos y responder al tratamiento el tiempo suficiente como para llegar al momento en que sea posible la administración de la terapia de células madre, no pueden presentar toxicidad comórbida que evite la terapia de dosis altas y, además, deben tener una adecuada recolección de células madre. Los pacientes que se someten a terapia de dosis altas y apoyo de células madre son un grupo muy seleccionado. La comparación de este grupo de pacientes con todos los pacientes con sarcoma de Ewing de riesgo alto es inadecuada y lleva a la conclusión errónea de que esta estrategia mejora el desenlace.
En las encuestas de pacientes sometidos a trasplante de células madre hematopoyéticas (TCMH) alogénico para sarcoma de Ewing recidivante no se observó ninguna mejora de la SSC, en comparación con los pacientes sometidos a TCMH autógeno. Además, el TCMH alogénico se relacionó con una tasa de complicaciones más alta.[41,45,46]
Otros tratamientos
Otros tratamientos que se han estudiado para el sarcoma de Ewing recidivante son los siguientes:
- Terapia con anticuerpos monoclonales. Se notificó que los anticuerpos monoclonales contra el receptor del factor de crecimiento 1 similar a la insulina (IGF1R) producen respuestas objetivas en cerca del 10 % de los pacientes con sarcoma de Ewing recidivante metastásico.[47-50][Nivel de evidencia C3] En estos estudios, se indicó que el tiempo hasta la progresión fue más prolongado cuando se comparó con controles históricos. Se notificaron respuestas objetivas en estudios en los que se combinó el inhibidor mTOR temsirólimus con un anticuerpo contra IGF1R. En un ensayo de fase II de 10 pacientes tratados con un anticuerpo contra IGF1R y un inhibidor de CDK4/6, no se notificaron respuestas.[51] La estratificación mediante la expresión de IGF1R (detectada con prueba inmunohistoquímica) en uno de los estudios no predijo el desenlace clínico de pacientes con sarcoma de Ewing.[52,53] Se necesitan más estudios con el fin de identificar a los pacientes que tienen más probabilidades de beneficiarse de la terapia con IGF1R.
- Inmunoterapia. La inmunoterapia con células T específicas para antígeno está en estudio en pacientes con sarcoma de Ewing porque la destrucción por mediación inmunitaria ataca el tumor de una manera diferente, a diferencia de las terapias convencionales (que dependen de vías), a las que estos tumores a menudo presentan resistencia. Se han identificado varios antígenos a los que se podrían dirigir receptores de antígenos quiméricos en los sarcomas de Ewing, que incluyen HER2 (receptor 2 del factor de crecimiento epidérmico humano),[54] GD2,[55] CD99 (antígenos MIC2),[56] y STEAP1 (6 antígenos epiteliales transmembrana de la próstata).[57] Algunas de estas terapias están en una fase temprana de investigación en pacientes con sarcomas.[54]
El tratamiento con inhibidores de puntos de control inmunitario en monoterapia y combinados no mostró actividad en pacientes con sarcoma de Ewing recidivante. No hubo respuestas en 5 pacientes con sarcoma de Ewing tratados en un ensayo clínico con nivolumab en monoterapia o con la combinación de nivolumab e ipilimumab.[58] En un ensayo de pembrolizumab se incluyeron 13 pacientes con sarcoma de Ewing cuyo cáncer no respondió al tratamiento.[59] En otro ensayo de nivolumab, se notificó que no hubo respuestas en 11 pacientes con sarcoma de Ewing.[60] El Children's Oncology Group (COG) llevó a cabo un ensayo de fase I/II de la combinación de nivolumab e ipilimumab en niños con sarcomas recidivantes.[61][Nivel de evidencia B4] Solo 1 de 14 pacientes con sarcoma de Ewing presentó una respuesta parcial prolongada.
-
Inhibidores de cinasas multidirigidos.
- Regorafenib: en un estudio de fase II (SARC024 [NCT02048371]) se evaluó la monoterapia con regorafenib en 30 pacientes con sarcoma de Ewing metastásico recidivante y otros tumores positivos para translocación, incluso el sarcoma con la translocación CIC::DUX4.[62] Se observó una actividad modesta, con una tasa de respuesta objetiva del 10 % según los Criterios de Evaluación de Respuesta en Tumores Sólidos (RECIST). La mediana de SSP fue de 14,8 semanas.
En un ensayo prospectivo aleatorizado con enmascaramiento doble, se comparó el regorafenib con placebo en pacientes con sarcoma de Ewing recidivante.[63] De 36 pacientes evaluables para la eficacia, 23 recibieron regorafenib y 13 placebo. Los pacientes asignados al azar a recibir regorafenib tuvieron una tasa de SSP a 8 semanas del 56 %, en comparación con el 7,7 % de los pacientes asignados al azar a recibir placebo. La mediana de SSP fue de 11,4 semanas en los pacientes que recibieron regorafenib y de 3,9 semanas en los pacientes que recibieron placebo. La tasa de respuesta fue del 13 % en los pacientes tratados con regorafenib. Del grupo de placebo, 10 pacientes pasaron a recibir regorafenib después de la progresión. Aunque los resultados no fueron significativos, los autores indicaron que este ensayo proporciona evidencia del beneficio del uso de regorafenib en pacientes con sarcoma de Ewing recidivante.
Además de la experiencia con el regorafenib en monoterapia, este fármaco se toleró en un programa de administración secuencial con vincristina e irinotecán en 21 pacientes pediátricos. En este ensayo de fase I se inscribieron 5 pacientes con sarcoma de Ewing en recaída. Se presentaron respuestas parciales en 3 de 5 pacientes que recibieron este régimen.[64]
- Anlotinib: en un estudio que se llevó a cabo en China, se evaluó el tratamiento con anlotinib (inhibidor de receptor de tirosina–cinasas oral dirigido a los receptores del factor de crecimiento endotelial vascular 1–3, factores de crecimiento de fibroblastos 1–3, receptores α y β del factor de crecimiento derivado de plaquetas, c-KIT y RET) administrado con vincristina e irinotecán para pacientes con sarcoma de Ewing en recaída o resistente al tratamiento.[65][Nivel de evidencia B4] Los pacientes se trataron en 2 cohortes. En la cohorte A se incluyeron pacientes de 16 años y más, y en la cohorte B participaron pacientes de menos de 16 años. A las 12 semanas, los pacientes en la cohorte A demostraron una tasa de respuesta objetiva del 62,5 % (14 de 23 pacientes) y los pacientes en la cohorte B demostraron una tasa de respuesta objetiva del 83,3 % (11 de 12 pacientes). La Administración de Alimentos y Medicamentos de los Estados Unidos todavía no ha otorgado la aprobación al anlotinib.
- Cabozantinib: en un análisis de evidencia de la vida real se incluyeron 16 pacientes con sarcoma de Ewing en recaída o resistente al tratamiento que recibieron cabozantinib. De los pacientes, 4 presentaron respuestas objetivas (tasa de respuesta objetiva del 25 %).[66]
Se evaluó la respuesta de 39 pacientes (mayores de 12 años) con sarcoma de Ewing en recaída después de la monoterapia con cabozantinib.[67] Los pacientes menores de 16 años recibieron 40 mg diarios y los pacientes de 16 años o más recibieron 60 mg diarios. La mayoría de los pacientes eran mayores de 18 años. De los pacientes, 10 (26 %) presentaron respuestas (todas las respuestas fueron parciales).
- Regorafenib: en un estudio de fase II (SARC024 [NCT02048371]) se evaluó la monoterapia con regorafenib en 30 pacientes con sarcoma de Ewing metastásico recidivante y otros tumores positivos para translocación, incluso el sarcoma con la translocación CIC::DUX4.[62] Se observó una actividad modesta, con una tasa de respuesta objetiva del 10 % según los Criterios de Evaluación de Respuesta en Tumores Sólidos (RECIST). La mediana de SSP fue de 14,8 semanas.
-
Estrategias dirigidas a las proteínas de fusión.
- Lurbinectedina: la lurbinectedina se relaciona de forma estructural con la trabectedina, pero ha sido más eficaz para suprimir la actividad del factor de transcripción oncogénico EWS::FLI en ratones en estudios preclínicos. En un ensayo en canasta sin enmascaramiento de fase II de un solo grupo, se observó actividad clínica antitumoral. En una cohorte de 28 pacientes adultos con sarcoma de Ewing confirmado y enfermedad en recaída, la tasa de respuesta objetiva fue del 14,3 %, la tasa de beneficio clínico (respuesta o estabilización de la enfermedad durante >4 meses) fue del 39,3 % y la tasa de control de la enfermedad (respuesta o estabilización de la enfermedad de cualquier duración) fue del 57,1 %.[68]
- TK216: se cree que un fármaco llamado TK216 interfiere con las interacciones entre la oncoproteína de fusión EWSR1::FLI1 y proteínas clave críticas para su funcionamiento oncogénico, aunque también se ha demostrado que altera los microtúbulos.[69] En un ensayo de fase I/II de TK216 en combinación con vincristina en pacientes con sarcoma de Ewing recidivante, se observaron 3 respuestas. Sin embargo, la tasa de respuesta general fue de solo el 3,5 % en todo el ensayo.[70]
La secuenciación de tumores de sarcoma de Ewing recidivantes y resistentes al tratamiento de pacientes pediátricos (n = 79) y adultos jóvenes (n = 25) inscritos en el ensayo National Cancer Institute (NCI)-COG Pediatric Molecular Analysis for Therapeutic Choice (MATCH) reveló alteraciones genómicas que se consideraron susceptibles de tratamiento en los grupos del estudio MATCH en 8 de 104 tumores (7,7 %), incluso las variantes de EZH2 en 2 de 104 tumores (1,9 %).[71]
Opciones de tratamiento en evaluación clínica para el sarcoma de Ewing recidivante
La información en inglés sobre los ensayos clínicos patrocinados por el NCI se encuentra en el portal de Internet del NCI. Para obtener información en inglés sobre ensayos clínicos patrocinados por otras organizaciones, consultar el portal de Internet ClinicalTrials.gov.
A continuación, se presenta un ejemplo de un ensayo clínico nacional o institucional en curso:
- NCT04890093 (Vincristine and Temozolomide in Combination With PEN-866 for Adolescents and Young Adults With Relapsed or Refractory Solid Tumors): la PEN-866 es una molécula novedosa que consiste en un conjugado de SN-38 con un inhibidor de la proteína de choque térmico 90 (HSP90) que se ha demostrado que tiene una ventaja farmacocinética sobre el irinotecán en modelos preclínicos. En los modelos preclínicos del sarcoma de Ewing, la PEN-866 tuvo una eficacia y una farmacodinámica superiores, en comparación con el irinotecán. Para la fase I de este ensayo, son aptos los pacientes de 12 a 39 años con tumor sólido en recaída o resistente al tratamiento. En la fase II de este ensayo solo se inscribirán pacientes de 12 a 39 años con sarcoma de Ewing o rabdomiosarcoma en recaída o resistente al tratamiento.
Ensayos clínicos en curso
Realizar una búsqueda avanzada en inglés de los ensayos clínicos sobre cáncer auspiciados por el NCI que ahora aceptan pacientes. La búsqueda se puede simplificar por ubicación del ensayo, tipo de tratamiento, nombre del fármaco y otros criterios. También se dispone de información general sobre los ensayos clínicos.
Bibliografía
- Leavey PJ, Mascarenhas L, Marina N, et al.: Prognostic factors for patients with Ewing sarcoma (EWS) at first recurrence following multi-modality therapy: A report from the Children's Oncology Group. Pediatr Blood Cancer 51 (3): 334-8, 2008. [PUBMED Abstract]
- Stahl M, Ranft A, Paulussen M, et al.: Risk of recurrence and survival after relapse in patients with Ewing sarcoma. Pediatr Blood Cancer 57 (4): 549-53, 2011. [PUBMED Abstract]
- Wasilewski-Masker K, Liu Q, Yasui Y, et al.: Late recurrence in pediatric cancer: a report from the Childhood Cancer Survivor Study. J Natl Cancer Inst 101 (24): 1709-20, 2009. [PUBMED Abstract]
- Davenport JR, Vo KT, Goldsby R, et al.: Conditional Survival and Predictors of Late Death in Patients With Ewing Sarcoma. Pediatr Blood Cancer 63 (6): 1091-5, 2016. [PUBMED Abstract]
- Barker LM, Pendergrass TW, Sanders JE, et al.: Survival after recurrence of Ewing's sarcoma family of tumors. J Clin Oncol 23 (19): 4354-62, 2005. [PUBMED Abstract]
- Bacci G, Longhi A, Ferrari S, et al.: Pattern of relapse in 290 patients with nonmetastatic Ewing's sarcoma family tumors treated at a single institution with adjuvant and neoadjuvant chemotherapy between 1972 and 1999. Eur J Surg Oncol 32 (9): 974-9, 2006. [PUBMED Abstract]
- Collier AB, Krailo MD, Dang HM, et al.: Outcome of patients with relapsed or progressive Ewing sarcoma enrolled on cooperative group phase 2 clinical trials: A report from the Children's Oncology Group. Pediatr Blood Cancer 68 (12): e29333, 2021. [PUBMED Abstract]
- Ferrari S, Luksch R, Hall KS, et al.: Post-relapse survival in patients with Ewing sarcoma. Pediatr Blood Cancer 62 (6): 994-9, 2015. [PUBMED Abstract]
- Saylors RL, Stine KC, Sullivan J, et al.: Cyclophosphamide plus topotecan in children with recurrent or refractory solid tumors: a Pediatric Oncology Group phase II study. J Clin Oncol 19 (15): 3463-9, 2001. [PUBMED Abstract]
- McTiernan A, Driver D, Michelagnoli MP, et al.: High dose chemotherapy with bone marrow or peripheral stem cell rescue is an effective treatment option for patients with relapsed or progressive Ewing's sarcoma family of tumours. Ann Oncol 17 (8): 1301-5, 2006. [PUBMED Abstract]
- Hunold A, Weddeling N, Paulussen M, et al.: Topotecan and cyclophosphamide in patients with refractory or relapsed Ewing tumors. Pediatr Blood Cancer 47 (6): 795-800, 2006. [PUBMED Abstract]
- Wagner LM, McAllister N, Goldsby RE, et al.: Temozolomide and intravenous irinotecan for treatment of advanced Ewing sarcoma. Pediatr Blood Cancer 48 (2): 132-9, 2007. [PUBMED Abstract]
- Casey DA, Wexler LH, Merchant MS, et al.: Irinotecan and temozolomide for Ewing sarcoma: the Memorial Sloan-Kettering experience. Pediatr Blood Cancer 53 (6): 1029-34, 2009. [PUBMED Abstract]
- Raciborska A, Bilska K, Drabko K, et al.: Vincristine, irinotecan, and temozolomide in patients with relapsed and refractory Ewing sarcoma. Pediatr Blood Cancer 60 (10): 1621-5, 2013. [PUBMED Abstract]
- Farhat R, Raad R, Khoury NJ, et al.: Cyclophosphamide and topotecan as first-line salvage therapy in patients with relapsed ewing sarcoma at a single institution. J Pediatr Hematol Oncol 35 (5): 356-60, 2013. [PUBMED Abstract]
- Kebudi R, Cakir FB, Gorgun O, et al.: A modified protocol with vincristine, topotecan, and cyclophosphamide for recurrent/progressive ewing sarcoma family tumors. Pediatr Hematol Oncol 30 (3): 170-7, 2013. [PUBMED Abstract]
- Hernández-Marqués C, Lassaletta-Atienza A, Ruiz Hernández A, et al.: [Irinotecan plus temozolomide in refractory or relapsed pediatric solid tumors]. An Pediatr (Barc) 79 (2): 68-74, 2013. [PUBMED Abstract]
- McNall-Knapp RY, Williams CN, Reeves EN, et al.: Extended phase I evaluation of vincristine, irinotecan, temozolomide, and antibiotic in children with refractory solid tumors. Pediatr Blood Cancer 54 (7): 909-15, 2010. [PUBMED Abstract]
- Wagner LM, Perentesis JP, Reid JM, et al.: Phase I trial of two schedules of vincristine, oral irinotecan, and temozolomide (VOIT) for children with relapsed or refractory solid tumors: a Children's Oncology Group phase I consortium study. Pediatr Blood Cancer 54 (4): 538-45, 2010. [PUBMED Abstract]
- Wagner L, Turpin B, Nagarajan R, et al.: Pilot study of vincristine, oral irinotecan, and temozolomide (VOIT regimen) combined with bevacizumab in pediatric patients with recurrent solid tumors or brain tumors. Pediatr Blood Cancer 60 (9): 1447-51, 2013. [PUBMED Abstract]
- Bagatell R, Norris R, Ingle AM, et al.: Phase 1 trial of temsirolimus in combination with irinotecan and temozolomide in children, adolescents and young adults with relapsed or refractory solid tumors: a Children's Oncology Group Study. Pediatr Blood Cancer 61 (5): 833-9, 2014. [PUBMED Abstract]
- Kurucu N, Sari N, Ilhan IE: Irinotecan and temozolamide treatment for relapsed Ewing sarcoma: a single-center experience and review of the literature. Pediatr Hematol Oncol 32 (1): 50-9, 2015. [PUBMED Abstract]
- Anderson P, Kopp L, Anderson N, et al.: Novel bone cancer drugs: investigational agents and control paradigms for primary bone sarcomas (Ewing's sarcoma and osteosarcoma). Expert Opin Investig Drugs 17 (11): 1703-15, 2008. [PUBMED Abstract]
- Palmerini E, Jones RL, Setola E, et al.: Irinotecan and temozolomide in recurrent Ewing sarcoma: an analysis in 51 adult and pediatric patients. Acta Oncol 57 (7): 958-964, 2018. [PUBMED Abstract]
- Salah S, To YH, Khozouz O, et al.: Irinotecan and temozolomide chemotherapy in paediatric and adult populations with relapsed Ewing Sarcoma. Clin Transl Oncol 23 (4): 757-763, 2021. [PUBMED Abstract]
- Xu J, Xie L, Sun X, et al.: Longer versus Shorter Schedules of Vincristine, Irinotecan, and Temozolomide (VIT) for Relapsed or Refractory Ewing Sarcoma: A Randomized Controlled Phase 2 Trial. Clin Cancer Res 29 (6): 1040-1046, 2023. [PUBMED Abstract]
- Slotkin EK, Meyers PA: Irinotecan dose schedule for the treatment of Ewing sarcoma. Pediatr Blood Cancer 70 (1): e30005, 2023. [PUBMED Abstract]
- Fox E, Patel S, Wathen JK, et al.: Phase II study of sequential gemcitabine followed by docetaxel for recurrent Ewing sarcoma, osteosarcoma, or unresectable or locally recurrent chondrosarcoma: results of Sarcoma Alliance for Research Through Collaboration Study 003. Oncologist 17 (3): 321, 2012. [PUBMED Abstract]
- Mora J, Cruz CO, Parareda A, et al.: Treatment of relapsed/refractory pediatric sarcomas with gemcitabine and docetaxel. J Pediatr Hematol Oncol 31 (10): 723-9, 2009. [PUBMED Abstract]
- Yoon JH, Kwon MM, Park HJ, et al.: A study of docetaxel and irinotecan in children and young adults with recurrent or refractory Ewing sarcoma family of tumors. BMC Cancer 14: 622, 2014. [PUBMED Abstract]
- Ferrari S, del Prever AB, Palmerini E, et al.: Response to high-dose ifosfamide in patients with advanced/recurrent Ewing sarcoma. Pediatr Blood Cancer 52 (5): 581-4, 2009. [PUBMED Abstract]
- McCabe MG, Moroz V, Khan M, et al.: Results of the first interim assessment of rEECur, an international randomized controlled trial of chemotherapy for the treatment of recurrent and primary refractory Ewing sarcoma. [Abstract] J Clin Oncol 37 (Suppl 15): A-11007, 2019. Also available online. Last accessed June 29, 2021.
- McCabe MG, Kirton L, Khan M, et al.: Results of the second interim assessment of rEECur, an international randomized controlled trial of chemotherapy for the treatment of recurrent and primary refractory Ewing sarcoma (RR-ES). [Abstract] J Clin Oncol 38 (Suppl 15): A-11502, 2020. Also available online. Last accessed June 29, 2021.
- Strauss SJ, Berlanga P, McCabe MG: Emerging therapies in Ewing sarcoma. Curr Opin Oncol 36 (4): 297-304, 2024. [PUBMED Abstract]
- Gerrand C, Bate J, Seddon B, et al.: Seeking international consensus on approaches to primary tumour treatment in Ewing sarcoma. Clin Sarcoma Res 10 (1): 21, 2020. [PUBMED Abstract]
- Rodriguez-Galindo C, Billups CA, Kun LE, et al.: Survival after recurrence of Ewing tumors: the St Jude Children's Research Hospital experience, 1979-1999. Cancer 94 (2): 561-9, 2002. [PUBMED Abstract]
- Briccoli A, Rocca M, Ferrari S, et al.: Surgery for lung metastases in Ewing's sarcoma of bone. Eur J Surg Oncol 30 (1): 63-7, 2004. [PUBMED Abstract]
- Scobioala S, Ranft A, Wolters H, et al.: Impact of Whole Lung Irradiation on Survival Outcome in Patients With Lung Relapsed Ewing Sarcoma. Int J Radiat Oncol Biol Phys 102 (3): 584-592, 2018. [PUBMED Abstract]
- Liu KX, Chen YH, Kozono D, et al.: Phase I/II Study of Stereotactic Body Radiation Therapy for Pulmonary Metastases in Pediatric Patients. Adv Radiat Oncol 5 (6): 1267-1273, 2020 Nov-Dec. [PUBMED Abstract]
- Sudmeier LJ, Madden N, Zhang C, et al.: Palliative radiotherapy for children: Symptom response and treatment-associated toxicity according to radiation therapy dose and fractionation. Pediatr Blood Cancer 70 (4): e30195, 2023. [PUBMED Abstract]
- Burdach S, van Kaick B, Laws HJ, et al.: Allogeneic and autologous stem-cell transplantation in advanced Ewing tumors. An update after long-term follow-up from two centers of the European Intergroup study EICESS. Stem-Cell Transplant Programs at Düsseldorf University Medical Center, Germany and St. Anna Kinderspital, Vienna, Austria. Ann Oncol 11 (11): 1451-62, 2000. [PUBMED Abstract]
- Burdach S, Meyer-Bahlburg A, Laws HJ, et al.: High-dose therapy for patients with primary multifocal and early relapsed Ewing's tumors: results of two consecutive regimens assessing the role of total-body irradiation. J Clin Oncol 21 (16): 3072-8, 2003. [PUBMED Abstract]
- Pawlowska AB, Sun V, Calvert GT, et al.: Long-Term Follow-up of High-Dose Chemotherapy with Autologous Stem Cell Transplantation in Children and Young Adults with Metastatic or Relapsed Ewing Sarcoma: A Single-Institution Experience. Transplant Cell Ther 27 (1): 72.e1-72.e7, 2021. [PUBMED Abstract]
- Gardner SL, Carreras J, Boudreau C, et al.: Myeloablative therapy with autologous stem cell rescue for patients with Ewing sarcoma. Bone Marrow Transplant 41 (10): 867-72, 2008. [PUBMED Abstract]
- Gilman AL, Oesterheld J: Myeloablative chemotherapy with autologous stem cell rescue for Ewing sarcoma. Bone Marrow Transplant 42 (11): 761; author reply 763, 2008. [PUBMED Abstract]
- Eapen M: Response to Dr Gilman. Bone Marrow Transplant 42 (11): 763, 2008.
- Malempati S, Weigel B, Ingle AM, et al.: Phase I/II trial and pharmacokinetic study of cixutumumab in pediatric patients with refractory solid tumors and Ewing sarcoma: a report from the Children's Oncology Group. J Clin Oncol 30 (3): 256-62, 2012. [PUBMED Abstract]
- Juergens H, Daw NC, Geoerger B, et al.: Preliminary efficacy of the anti-insulin-like growth factor type 1 receptor antibody figitumumab in patients with refractory Ewing sarcoma. J Clin Oncol 29 (34): 4534-40, 2011. [PUBMED Abstract]
- Pappo AS, Patel SR, Crowley J, et al.: R1507, a monoclonal antibody to the insulin-like growth factor 1 receptor, in patients with recurrent or refractory Ewing sarcoma family of tumors: results of a phase II Sarcoma Alliance for Research through Collaboration study. J Clin Oncol 29 (34): 4541-7, 2011. [PUBMED Abstract]
- Tap WD, Demetri G, Barnette P, et al.: Phase II study of ganitumab, a fully human anti-type-1 insulin-like growth factor receptor antibody, in patients with metastatic Ewing family tumors or desmoplastic small round cell tumors. J Clin Oncol 30 (15): 1849-56, 2012. [PUBMED Abstract]
- Shulman DS, Merriam P, Choy E, et al.: Phase 2 trial of palbociclib and ganitumab in patients with relapsed Ewing sarcoma. Cancer Med 12 (14): 15207-15216, 2023. [PUBMED Abstract]
- Naing A, LoRusso P, Fu S, et al.: Insulin growth factor-receptor (IGF-1R) antibody cixutumumab combined with the mTOR inhibitor temsirolimus in patients with refractory Ewing's sarcoma family tumors. Clin Cancer Res 18 (9): 2625-31, 2012. [PUBMED Abstract]
- Schwartz GK, Tap WD, Qin LX, et al.: Cixutumumab and temsirolimus for patients with bone and soft-tissue sarcoma: a multicentre, open-label, phase 2 trial. Lancet Oncol 14 (4): 371-82, 2013. [PUBMED Abstract]
- Ahmed N, Brawley VS, Hegde M, et al.: Human Epidermal Growth Factor Receptor 2 (HER2) -Specific Chimeric Antigen Receptor-Modified T Cells for the Immunotherapy of HER2-Positive Sarcoma. J Clin Oncol 33 (15): 1688-96, 2015. [PUBMED Abstract]
- Pule MA, Savoldo B, Myers GD, et al.: Virus-specific T cells engineered to coexpress tumor-specific receptors: persistence and antitumor activity in individuals with neuroblastoma. Nat Med 14 (11): 1264-70, 2008. [PUBMED Abstract]
- Scotlandi K, Baldini N, Cerisano V, et al.: CD99 engagement: an effective therapeutic strategy for Ewing tumors. Cancer Res 60 (18): 5134-42, 2000. [PUBMED Abstract]
- Grunewald TG, Diebold I, Esposito I, et al.: STEAP1 is associated with the invasive and oxidative stress phenotype of Ewing tumors. Mol Cancer Res 10 (1): 52-65, 2012. [PUBMED Abstract]
- D'Angelo SP, Mahoney MR, Van Tine BA, et al.: Nivolumab with or without ipilimumab treatment for metastatic sarcoma (Alliance A091401): two open-label, non-comparative, randomised, phase 2 trials. Lancet Oncol 19 (3): 416-426, 2018. [PUBMED Abstract]
- Tawbi HA, Burgess M, Bolejack V, et al.: Pembrolizumab in advanced soft-tissue sarcoma and bone sarcoma (SARC028): a multicentre, two-cohort, single-arm, open-label, phase 2 trial. Lancet Oncol 18 (11): 1493-1501, 2017. [PUBMED Abstract]
- Davis KL, Fox E, Merchant MS, et al.: Nivolumab in children and young adults with relapsed or refractory solid tumours or lymphoma (ADVL1412): a multicentre, open-label, single-arm, phase 1-2 trial. Lancet Oncol 21 (4): 541-550, 2020. [PUBMED Abstract]
- Davis KL, Fox E, Isikwei E, et al.: A Phase I/II Trial of Nivolumab plus Ipilimumab in Children and Young Adults with Relapsed/Refractory Solid Tumors: A Children's Oncology Group Study ADVL1412. Clin Cancer Res 28 (23): 5088-5097, 2022. [PUBMED Abstract]
- Attia S, Bolejack V, Ganjoo KN, et al.: A phase II trial of regorafenib in patients with advanced Ewing sarcoma and related tumors of soft tissue and bone: SARC024 trial results. Cancer Med 12 (2): 1532-1539, 2023. [PUBMED Abstract]
- Duffaud F, Blay JY, Le Cesne A, et al.: Regorafenib in patients with advanced Ewing sarcoma: results of a non-comparative, randomised, double-blind, placebo-controlled, multicentre Phase II study. Br J Cancer 129 (12): 1940-1948, 2023. [PUBMED Abstract]
- Casanova M, Bautista F, Campbell-Hewson Q, et al.: Regorafenib plus Vincristine and Irinotecan in Pediatric Patients with Recurrent/Refractory Solid Tumors: An Innovative Therapy for Children with Cancer Study. Clin Cancer Res 29 (21): 4341-4351, 2023. [PUBMED Abstract]
- Xu J, Xie L, Sun X, et al.: Anlotinib, Vincristine, and Irinotecan for Advanced Ewing Sarcoma After Failure of Standard Multimodal Therapy: A Two-Cohort, Phase Ib/II Trial. Oncologist 26 (7): e1256-e1262, 2021. [PUBMED Abstract]
- Peretz Soroka H, Vora T, Noujaim J, et al.: Real-world experience of tyrosine kinase inhibitors in children, adolescents and adults with relapsed or refractory bone tumours: A Canadian Sarcoma Research and Clinical Collaboration (CanSaRCC) study. Cancer Med 12 (18): 18872-18881, 2023. [PUBMED Abstract]
- Italiano A, Mir O, Mathoulin-Pelissier S, et al.: Cabozantinib in patients with advanced Ewing sarcoma or osteosarcoma (CABONE): a multicentre, single-arm, phase 2 trial. Lancet Oncol 21 (3): 446-455, 2020. [PUBMED Abstract]
- Subbiah V, Braña I, Longhi A, et al.: Antitumor Activity of Lurbinectedin, a Selective Inhibitor of Oncogene Transcription, in Patients with Relapsed Ewing Sarcoma: Results of a Basket Phase II Study. Clin Cancer Res 28 (13): 2762-2770, 2022. [PUBMED Abstract]
- Povedano JM, Li V, Lake KE, et al.: TK216 targets microtubules in Ewing sarcoma cells. Cell Chem Biol 29 (8): 1325-1332.e4, 2022. [PUBMED Abstract]
- Meyers PA, Federman N, Daw N, et al.: Open-Label, Multicenter, Phase I/II, First-in-Human Trial of TK216: A First-Generation EWS::FLI1 Fusion Protein Antagonist in Ewing Sarcoma. J Clin Oncol 42 (31): 3725-3734, 2024. [PUBMED Abstract]
- Parsons DW, Janeway KA, Patton DR, et al.: Actionable Tumor Alterations and Treatment Protocol Enrollment of Pediatric and Young Adult Patients With Refractory Cancers in the National Cancer Institute-Children's Oncology Group Pediatric MATCH Trial. J Clin Oncol 40 (20): 2224-2234, 2022. [PUBMED Abstract]
Sarcomas indiferenciados de células redondas pequeñas (similares al Ewing)
Hay sarcomas indiferenciados de células redondas pequeñas de hueso y tejido blando que no tienen las translocaciones del gen EWSR1 ni un gen de la familia ETS. Estos sarcomas se distinguen desde el punto de vista biológico del sarcoma de Ewing con translocaciones del gen EWSR1 y de genes de la familia ETS. Esto incluye los tumores con translocaciones del gen CIC o el gen BCOR, así como tumores con translocaciones de EWSR1 con genes que no pertenecen a la familia de genes ETS. Este grupo de tumores es mucho menos frecuente que el sarcoma de Ewing y los desenlaces clínicos definitivos para estos pacientes se basan en muestras más pequeñas y un tratamiento menos homogéneo; por consiguiente, es difícil cuantificar con precisión los desenlaces de los pacientes. La mayoría de estos tumores se han tratado con regímenes diseñados para el sarcoma de Ewing y el consenso es que, en el pasado, estos casos a menudo se incluyeron en ensayos clínicos para el tratamiento del sarcoma de Ewing, a veces llamado sarcoma de Ewing negativo para translocaciones. Ahora se acepta que estos tumores se diferencian lo suficiente del sarcoma de Ewing y, por lo tanto, se deben estadificar y analizar de forma independiente del sarcoma de Ewing con translocaciones comunes, aunque el tratamiento sea similar. A continuación, se presenta una descripción breve de estas entidades según el sistema de clasificación de la Organización Mundial de la Salud (OMS) de 2020, Classification of Tumours: Soft Tissue and Bone Tumours (5.ª edición).[1]
Sarcomas indiferenciados de células redondas pequeñas con alteraciones genéticas en BCOR
Cuadro clínico inicial
Los sarcomas indiferenciados de células redondas con reordenamientos BCOR::CCNB3 representan alrededor del 5 % de todos los sarcomas negativos para reordenamientos de EWSR1 y suelen afectar a varones. Más del 70 % de los casos se encuentran en pacientes menores de 18 años (mediana de edad en el momento del diagnóstico, 13–15 años).[2,3][Nivel de evidencia C1] Estos tumores aparecen con mayor frecuencia en los huesos de la pelvis y las extremidades, y se observan metástasis en alrededor del 30 % de los casos.
Características genómicas
Los tipos más comunes de sarcoma indiferenciado de células redondas pequeñas con reordenamientos BCOR son aquellos con el reordenamiento BCOR::CCNB3.[2,4] El reordenamiento BCOR::MAML3 se observa con menos frecuencia, pero los tumores con esta translocación tienen características biológicas similares a los tumores con el reordenamiento BCOR::CCNB3.[2,5,6]
Se observan duplicaciones internas en tándem (ITD) en BCOR que afectan el exón 15 en los sarcomas indiferenciados de células redondas infantiles y los tumores mesenquimatosos mixoides primitivos de la infancia (PMMTI).[7-9] Estas dos entidades coinciden en muchas características histológicas y en el perfil de transcripción, pero se diferencian por un estroma mixoide más abundante en el caso de PMMTI. A veces, se observan ITD en BCOR en los sarcomas indiferenciados de células redondas que se presentan en niños más grandes.[9]
Se notificaron ITD en BCOR en el 90 % de los casos de sarcoma de células claras de riñón, y un subconjunto más pequeño alberga las fusiones génicas YWHAE::NUTM2B/E o BCOR::CCNB3.[10,11] Para obtener más información, consulte la sección Sarcoma de células claras de riñón en Tratamiento del tumor de Wilms y otros tumores renales infantiles.
Los perfiles de transcripción inducidos por las fusiones génicas de BCOR, las ITD en BCOR y las fusiones YWHAE::NUTM2B/E son similares entre sí y distintos de los del sarcoma de Ewing.[2,6,7] Por ejemplo, en todas estas entidades se observa una expresión elevada de BCOR, lo que quizás sea útil para diferenciar estas entidades de otros tipos de tumores indiferenciados de células redondas pequeñas.
Tratamiento de los sarcomas indiferenciados de células redondas con alteraciones genéticas en BCOR
El 75 % de los pacientes exhiben respuestas patológicas significativas relacionadas con el tratamiento cuando reciben tratamientos indicados para el sarcoma de Ewing. En una serie de 36 casos, la tasa de supervivencia a 3 y 5 años, fue del 93 % y del 72 %, respectivamente.[2][Nivel de evidencia C1] En otra serie de 26 pacientes, la tasa de supervivencia general (SG) a 5 años fue del 76,5 % y la supervivencia fue superior en los pacientes que recibieron terapia de inducción con un régimen indicado para el sarcoma de Ewing.[12][Nivel de evidencia C1] La mayoría de los tumores de estas series surgieron en el hueso. En una encuesta retrospectiva de centros oncológicos europeos, se identificaron 148 pacientes con sarcomas indiferenciados de células redondas pequeñas que no tenían un gen de fusión relacionado con el sarcoma de Ewing.[13] De los 148 pacientes, 88 (60 %) tenían sarcomas con reordenamiento de CIC (mediana de edad, 32 años; intervalo 7–78 años), 33 (22 %) tenían sarcomas con el reordenamiento BCOR::CCNB3 (mediana de edad, 17 años; intervalo 5–91 años) y 27 (18 %) tenían sarcomas indiferenciados de células redondas pequeñas no clasificados (mediana de edad, 37 años; intervalo 4–70 años). De los 148 pacientes, 101 (68,2 %) presentaba enfermedad localizada y 47 (31,8 %) tenía metástasis en el momento del diagnóstico. Se observó una prevalencia de sexo masculino, edad más joven, sitio primario óseo y una tasa baja de metástasis sincrónicas en los casos con reordenamiento BCOR::CCNB3. El tratamiento local fue la cirugía para 67 pacientes (45 %) y cirugía con radioterapia para 52 pacientes (35 %). Se administró quimioterapia a 122 pacientes (82 %). Al cabo de una mediana de seguimiento de 42,7 meses, la tasa de SG a 3 años fue del 92,2 % en los pacientes con sarcomas con el reordenamiento BCOR::CCNB3, del 39,6 % en los pacientes con sarcomas con reordenamiento de CIC y del 78,7 % (P < 0,0001) en los pacientes con sarcomas indiferenciados de células redondas pequeñas no clasificados.
En un análisis retrospectivo multiinstitucional de pacientes de 0 a 24 años, se identificaron 29 pacientes con sarcomas y fusiones génicas CIC y 25 pacientes con sarcomas relacionados con BCOR (18 con fusiones génicas BCOR::CCNB3 y 7 con ITD en BCOR).[14] Al usar una amplia gama de tratamientos, las tasas de supervivencia sin complicaciones (SSC) a 3 años fueron del 44 % (intervalo de confianza [IC] 95 %, 28,7–67,5 %) para los pacientes con fusiones génicas de CIC y del 41,2 % (IC 95 %, 25,4–67 %) para los pacientes con alteraciones en BCOR (P = 0,97).
Sarcomas indiferenciados de células redondas pequeñas con alteraciones genéticas en CIC
Los sarcomas con reordenamiento de CIC representan la segunda familia más común de sarcomas de células redondas y se definen por la presencia de fusiones de CIC a nivel molecular.[1]
Cuadro clínico inicial
Los sarcomas indiferenciados de células redondas pequeñas con reordenamientos CIC::DUX4 suelen afectar a adultos jóvenes; el 50 % de los casos se presentan entre los 21 y los 40 años de edad. En una serie de 115 casos, la mediana de edad en el momento del diagnóstico fue de 32 años y el 22 % de los casos se presentaron en pacientes menores de 18 años.[3,15] Esta entidad por lo general afecta a varones y suele originarse en tejidos blandos del tronco y las extremidades.
Características genómicas
Los sarcomas con el reordenamiento de CIC suelen tener una fusión génica de CIC con DUX4, FOXO4 o NUTM1.[15-17] La fusión génica de CIC con DUX4 se debe a una translocación t(4;19)(q35;q13) o t(10;19)(q26;q13).[16,18] El gen CIC está en el cromosoma 19q13.1 y el gen DUX4 en el cromosoma 4q35 o 10q26.3. Los sarcomas con el reordenamiento CIC::DUX4 tienen un perfil de transcripción y un perfil de metilación del DNA que difieren del sarcoma de Ewing, lo que respalda su caracterización como una entidad distintiva.[6,19,20] Por ejemplo, casi todos los sarcomas con reordenamientos CIC::DUX4 expresan WT1 y ETV4, a diferencia del sarcoma de Ewing y los tumores con reordenamiento de BCOR, lo que hace que las pruebas inmunohistoquímicas para detectar estas proteínas sean útiles para distinguir entre estos diagnósticos.[15,19]
Tratamiento de los sarcomas indiferenciados de células redondas pequeñas con alteraciones genéticas en CIC
En una serie de 115 casos de sarcoma de células redondas pequeñas con reordenamiento de CIC, se obtuvo adecuada información de seguimiento para 57 pacientes.[15] De los pacientes, 9 presentaron metástasis y el 53 % de los pacientes con enfermedad localizada presentaron una recidiva que afectó con frecuencia el pulmón. Los pacientes que recibieron quimioterapia neoadyuvante tuvieron una supervivencia inferior a la de los pacientes sometidos a resección quirúrgica inicial. No obstante, es posible que esta diferencia en la supervivencia se deba a un tumor de mayor tamaño en el cuadro clínico inicial del primer grupo. Las tasas de supervivencia a 2 y 5 años fueron del 53 % y el 43 %, respectivamente.
En un estudio de cohorte retrospectivo internacional se destacó aún más los pronósticos precarios de los pacientes con sarcomas con reordenamientos de CIC. La tasa de SG a 3 años fue del 39 %, significativamente más precaria que los desenlaces de los pacientes con otros sarcomas indiferenciados de células redondas.[13] Del mismo modo, estas tasas de supervivencia son significativamente más bajas que las tasas de supervivencia observadas en pacientes con sarcoma de Ewing. Se necesitan más estudios para identificar el tratamiento óptimo de esta enfermedad.
En otra serie de 79 pacientes con sarcomas de células redondas con reordenamiento de CIC, los desenlaces fueron igualmente precarios, con una mediana de SG de 18 meses.[21] Los pacientes tratados con regímenes de quimioterapia para el sarcoma de Ewing presentaron tasas de respuesta nominalmente más altas, en comparación con los pacientes tratados con regímenes para el sarcoma de tejido blando. Sin embargo, las tasas de SG fueron similares en los 2 grupos.
En un análisis retrospectivo multiinstitucional de pacientes de 0 a 24 años, se identificaron 29 pacientes con sarcomas y fusiones génicas CIC y 25 pacientes con sarcomas relacionados con BCOR (18 con fusiones génicas BCOR::CCNB3 y 7 con ITD en BCOR).[14] Al usar una amplia gama de tratamientos, las tasas de SSC a 3 años fueron del 44 % (IC 95 %, 28,7–67,5 %) en los pacientes con fusiones génicas de CIC y del 41,2 % (IC 95 %, 25,4–67 %) en los pacientes con alteraciones en BCOR (P = 0,97).
Sarcomas indiferenciados de células redondas pequeñas con reordenamientos CIC::NUTM1
Los sarcomas indiferenciados de células redondas pequeñas con reordenamientos CIC::NUTM1 se presentan con mucha menos frecuencia que los sarcomas indiferenciados de células redondas con reordenamientos CIC::DUX4.[22-25] Estos tumores se presentan en pacientes más jóvenes, con una mediana de edad de 6 años, en comparación con una media de edad de 21,6 años para los pacientes con fusiones CIC::DUX4. Los tumores primarios se presentan en el sistema nervioso central (SNC) y en la periferia. El aspecto histológico de estos tumores es similar al de los sarcomas con reordenamientos CIC::DUX4. El pronóstico de los pacientes con estos tumores es muy precario a pesar del tratamiento con cirugía, quimioterapia multifarmacológica y radioterapia.
Sarcomas indiferenciado de células redondas pequeñas con fusiones ATXN1::NUTM2A o ATXN1L::NUTM2A
En un informe, 3 niños presentaban tumores con fusiones ATXN1::NUTM2A o ATXN1L::NUTM2A.[26] De los pacientes, 2 eran lactantes con lesiones en el SNC y el tercero era un neonato con compromiso cutáneo y masas múltiples en toda la cavidad peritoneal. Los autores indicaron que las fusiones que involucran los genes ATXN1 o ATXN1L desestabilizaron la interacción con CIC y redujeron el complejo represor de la transcripción con la consecuente sobreexpresión de la familia de genes PEA3 que se sitúa más adelante en la secuencia.
Sarcomas indiferenciados de células redondas pequeñas con fusiones de EWSR1 con genes que no pertenecen a la familia ETS
Sarcomas con fusiones EWSR1::NFATC2 y FUS::NFATC2
Por lo general, los sarcomas con fusiones EWSR1::NFATC2 y FUS::NFATC2 surgen en los huesos largos, muestran un fuerte predominio masculino y son más comunes en adultos que en niños.[27,28] Estas entidades tienen perfiles de transcripción y de metilación del DNA que las distinguen del sarcoma de Ewing y otros sarcomas de células redondas pequeñas.[6,20] Además, los perfiles de transcripción de EWSR1::NFATC2 y FUS::NFATC2 difieren entre sí,[6] aunque la importancia de esta observación no está clara. Las dos entidades también difieren en que se observa con frecuencia la amplificación del gen de fusión EWSR1::NFATC2, pero la fusión génica FUS::FNATC2, por lo general, no se amplifica.[20,27,29] Los sarcomas con fusiones EWSR1::NFATC2 y FUS::NFATC2 tienen potencial metastásico y no responden bien a los regímenes quimioterapéuticos que se usan con frecuencia para tratar otros sarcomas.[27,28]
También se observan reordenamientos EWSR1::NFATC2 y FUS::NFATC2 en una proporción importante de quistes óseos solitarios (también llamados quistes óseos simples), una afección benigna que se suele presentar en las metadiáfisis de los huesos largos del esqueleto inmaduro.[30,31] Por lo tanto, la presencia de fusiones EWSR1::NFATC2 o FUS::NFATC2 no se debe considerar indicador de neoplasia maligna, mas bien debe interpretarse en el contexto de los hallazgos clínicos.
Sarcomas con fusiones EWSR1::PATZ1
Los sarcomas con la fusión EWSR1::PATZ1 son muy infrecuentes. En el pequeño número de casos descritos, hay equilibrio de género, una propensión a la presentación en los sitios primarios troncales (en particular, el tórax) y una mediana de edad de presentación de entre 40 y 50 años; rara vez se presenta en la edad pediátrica.[32-34] Los sarcomas con la fusión EWSR1::PATZ1 tienen expresión génica y perfiles de metilación del DNA que los distinguen de otros sarcomas,[6,20] y las deleciones de CDKN2A se producen, por lo general, como alteraciones genómicas secundarias.[32,33]
La fusión EWSR1::PATZ1 se ha descrito de manera más frecuente en tumores encefálicos. Se ha indicado que esta fusión quizás defina un nuevo tipo de glioblastoma.[35] En una serie de 11 casos de tumores relacionados con la fusión EWSR1::PATZ1, 3 de ellos fueron tumores primarios de encéfalo, 7 fueron sarcomas y 1 se clasificó como sarcoma de tejido blando en el SNC.[32] Los pacientes tenían entre 11 y 81 años. Se notificó el tratamiento detallado solo para 3 adultos, 2 de ellos presentaron respuestas variadas a la quimioterapia seguidas de progresión de la enfermedad, y 1 paciente no recibió quimioterapia.
Bibliografía
- WHO Classification of Tumours Editorial Board: WHO Classification of Tumours. Volume 3: Soft Tissue and Bone Tumours. 5th ed., IARC Press, 2020.
- Kao YC, Owosho AA, Sung YS, et al.: BCOR-CCNB3 Fusion Positive Sarcomas: A Clinicopathologic and Molecular Analysis of 36 Cases With Comparison to Morphologic Spectrum and Clinical Behavior of Other Round Cell Sarcomas. Am J Surg Pathol 42 (5): 604-615, 2018. [PUBMED Abstract]
- Machado I, Navarro S, Llombart-Bosch A: Ewing sarcoma and the new emerging Ewing-like sarcomas: (CIC and BCOR-rearranged-sarcomas). A systematic review. Histol Histopathol 31 (11): 1169-81, 2016. [PUBMED Abstract]
- Pierron G, Tirode F, Lucchesi C, et al.: A new subtype of bone sarcoma defined by BCOR-CCNB3 gene fusion. Nat Genet 44 (4): 461-6, 2012. [PUBMED Abstract]
- Specht K, Zhang L, Sung YS, et al.: Novel BCOR-MAML3 and ZC3H7B-BCOR Gene Fusions in Undifferentiated Small Blue Round Cell Sarcomas. Am J Surg Pathol 40 (4): 433-42, 2016. [PUBMED Abstract]
- Watson S, Perrin V, Guillemot D, et al.: Transcriptomic definition of molecular subgroups of small round cell sarcomas. J Pathol 245 (1): 29-40, 2018. [PUBMED Abstract]
- Kao YC, Sung YS, Zhang L, et al.: Recurrent BCOR Internal Tandem Duplication and YWHAE-NUTM2B Fusions in Soft Tissue Undifferentiated Round Cell Sarcoma of Infancy: Overlapping Genetic Features With Clear Cell Sarcoma of Kidney. Am J Surg Pathol 40 (8): 1009-20, 2016. [PUBMED Abstract]
- Kao YC, Sung YS, Zhang L, et al.: BCOR Overexpression Is a Highly Sensitive Marker in Round Cell Sarcomas With BCOR Genetic Abnormalities. Am J Surg Pathol 40 (12): 1670-1678, 2016. [PUBMED Abstract]
- Antonescu CR, Kao YC, Xu B, et al.: Undifferentiated round cell sarcoma with BCOR internal tandem duplications (ITD) or YWHAE fusions: a clinicopathologic and molecular study. Mod Pathol 33 (9): 1669-1677, 2020. [PUBMED Abstract]
- Ueno-Yokohata H, Okita H, Nakasato K, et al.: Consistent in-frame internal tandem duplications of BCOR characterize clear cell sarcoma of the kidney. Nat Genet 47 (8): 861-3, 2015. [PUBMED Abstract]
- Roy A, Kumar V, Zorman B, et al.: Recurrent internal tandem duplications of BCOR in clear cell sarcoma of the kidney. Nat Commun 6: 8891, 2015. [PUBMED Abstract]
- Cohen-Gogo S, Cellier C, Coindre JM, et al.: Ewing-like sarcomas with BCOR-CCNB3 fusion transcript: a clinical, radiological and pathological retrospective study from the Société Française des Cancers de L'Enfant. Pediatr Blood Cancer 61 (12): 2191-8, 2014. [PUBMED Abstract]
- Palmerini E, Gambarotti M, Italiano A, et al.: A global collaboRAtive study of CIC-rearranged, BCOR::CCNB3-rearranged and other ultra-rare unclassified undifferentiated small round cell sarcomas (GRACefUl). Eur J Cancer 183: 11-23, 2023. [PUBMED Abstract]
- Sparber-Sauer M, Corradini N, Affinita MC, et al.: Clinical characteristics and outcomes for children, adolescents and young adults with "CIC-fused" or "BCOR-rearranged" soft tissue sarcomas: A multi-institutional European retrospective analysis. Cancer Med 12 (13): 14346-14359, 2023. [PUBMED Abstract]
- Antonescu CR, Owosho AA, Zhang L, et al.: Sarcomas With CIC-rearrangements Are a Distinct Pathologic Entity With Aggressive Outcome: A Clinicopathologic and Molecular Study of 115 Cases. Am J Surg Pathol 41 (7): 941-949, 2017. [PUBMED Abstract]
- Italiano A, Sung YS, Zhang L, et al.: High prevalence of CIC fusion with double-homeobox (DUX4) transcription factors in EWSR1-negative undifferentiated small blue round cell sarcomas. Genes Chromosomes Cancer 51 (3): 207-18, 2012. [PUBMED Abstract]
- Dickson BC, Sung YS, Rosenblum MK, et al.: NUTM1 Gene Fusions Characterize a Subset of Undifferentiated Soft Tissue and Visceral Tumors. Am J Surg Pathol 42 (5): 636-645, 2018. [PUBMED Abstract]
- Kawamura-Saito M, Yamazaki Y, Kaneko K, et al.: Fusion between CIC and DUX4 up-regulates PEA3 family genes in Ewing-like sarcomas with t(4;19)(q35;q13) translocation. Hum Mol Genet 15 (13): 2125-37, 2006. [PUBMED Abstract]
- Specht K, Sung YS, Zhang L, et al.: Distinct transcriptional signature and immunoprofile of CIC-DUX4 fusion-positive round cell tumors compared to EWSR1-rearranged Ewing sarcomas: further evidence toward distinct pathologic entities. Genes Chromosomes Cancer 53 (7): 622-33, 2014. [PUBMED Abstract]
- Koelsche C, Kriegsmann M, Kommoss FKF, et al.: DNA methylation profiling distinguishes Ewing-like sarcoma with EWSR1-NFATc2 fusion from Ewing sarcoma. J Cancer Res Clin Oncol 145 (5): 1273-1281, 2019. [PUBMED Abstract]
- Brahmi M, Gaspar N, Gantzer J, et al.: Patterns of care and outcome of CIC-rearranged sarcoma patients: A nationwide study of the French sarcoma group. Cancer Med 12 (7): 7801-7807, 2023. [PUBMED Abstract]
- Le Loarer F, Pissaloux D, Watson S, et al.: Clinicopathologic Features of CIC-NUTM1 Sarcomas, a New Molecular Variant of the Family of CIC-Fused Sarcomas. Am J Surg Pathol 43 (2): 268-276, 2019. [PUBMED Abstract]
- Mangray S, Kelly DR, LeGuellec S, et al.: Clinicopathologic Features of a Series of Primary Renal CIC-rearranged Sarcomas With Comprehensive Molecular Analysis. Am J Surg Pathol 42 (10): 1360-1369, 2018. [PUBMED Abstract]
- Schaefer IM, Dal Cin P, Landry LM, et al.: CIC-NUTM1 fusion: A case which expands the spectrum of NUT-rearranged epithelioid malignancies. Genes Chromosomes Cancer 57 (9): 446-451, 2018. [PUBMED Abstract]
- Zhao L, He H, Ren J, et al.: CIC::NUTM1 sarcomas occurred in soft tissues of upper limbs : a rare case report and literature review. Diagn Pathol 19 (1): 76, 2024. [PUBMED Abstract]
- Xu F, Viaene AN, Ruiz J, et al.: Novel ATXN1/ATXN1L::NUTM2A fusions identified in aggressive infant sarcomas with gene expression and methylation patterns similar to CIC-rearranged sarcoma. Acta Neuropathol Commun 10 (1): 102, 2022. [PUBMED Abstract]
- Bode-Lesniewska B, Fritz C, Exner GU, et al.: EWSR1-NFATC2 and FUS-NFATC2 Gene Fusion-Associated Mesenchymal Tumors: Clinicopathologic Correlation and Literature Review. Sarcoma 2019: 9386390, 2019. [PUBMED Abstract]
- Wang GY, Thomas DG, Davis JL, et al.: EWSR1-NFATC2 Translocation-associated Sarcoma Clinicopathologic Findings in a Rare Aggressive Primary Bone or Soft Tissue Tumor. Am J Surg Pathol 43 (8): 1112-1122, 2019. [PUBMED Abstract]
- Szuhai K, Ijszenga M, de Jong D, et al.: The NFATc2 gene is involved in a novel cloned translocation in a Ewing sarcoma variant that couples its function in immunology to oncology. Clin Cancer Res 15 (7): 2259-68, 2009. [PUBMED Abstract]
- Pižem J, Šekoranja D, Zupan A, et al.: FUS-NFATC2 or EWSR1-NFATC2 Fusions Are Present in a Large Proportion of Simple Bone Cysts. Am J Surg Pathol 44 (12): 1623-1634, 2020. [PUBMED Abstract]
- Hung YP, Fisch AS, Diaz-Perez JA, et al.: Identification of EWSR1-NFATC2 fusion in simple bone cysts. Histopathology 78 (6): 849-856, 2021. [PUBMED Abstract]
- Bridge JA, Sumegi J, Druta M, et al.: Clinical, pathological, and genomic features of EWSR1-PATZ1 fusion sarcoma. Mod Pathol 32 (11): 1593-1604, 2019. [PUBMED Abstract]
- Michal M, Rubin BP, Agaimy A, et al.: EWSR1-PATZ1-rearranged sarcoma: a report of nine cases of spindle and round cell neoplasms with predilection for thoracoabdominal soft tissues and frequent expression of neural and skeletal muscle markers. Mod Pathol 34 (4): 770-785, 2021. [PUBMED Abstract]
- Dehner CA, Torres-Mora J, Gupta S, et al.: Sarcomas Harboring EWSR1::PATZ1 Fusions: A Clinicopathologic Study of 17 Cases. Mod Pathol 37 (2): 100400, 2024. [PUBMED Abstract]
- Siegfried A, Rousseau A, Maurage CA, et al.: EWSR1-PATZ1 gene fusion may define a new glioneuronal tumor entity. Brain Pathol 29 (1): 53-62, 2019. [PUBMED Abstract]
Actualizaciones más recientes a este resumen (05/13/2025)
Los resúmenes del PDQ con información sobre el cáncer se revisan con regularidad y se actualizan a medida que se obtiene nueva información. Esta sección describe los cambios más recientes introducidos en este resumen a partir de la fecha arriba indicada.
Este resumen fue objeto de revisión integral.
El Consejo editorial del PDQ sobre el tratamiento pediátrico es responsable de la redacción y actualización de este resumen y mantiene independencia editorial respecto del NCI. El resumen refleja una revisión independiente de la bibliografía médica y no representa las políticas del NCI ni de los NIH. Para obtener más información sobre las políticas relativas a los resúmenes y la función de los consejos editoriales del PDQ responsables de su actualización, consultar Información sobre este resumen del PDQ e Información del PDQ® sobre el cáncer dirigida a profesionales de la salud.
Información sobre este resumen del PDQ
Propósito de este resumen
Este resumen de información del PDQ sobre el cáncer dirigido a profesionales de la salud proporciona información integral revisada por expertos y basada en la evidencia sobre el tratamiento del sarcoma de Ewing infantil y los sarcomas indiferenciados de células redondas pequeñas de hueso y tejido blando. El objetivo es servir como fuente de información y ayuda para los profesionales clínicos durante la atención de pacientes. No ofrece pautas ni recomendaciones formales para tomar decisiones relacionadas con la atención sanitaria.
Revisores y actualizaciones
El consejo editorial del PDQ sobre el tratamiento pediátrico, que mantiene independencia editorial respecto del Instituto Nacional del Cáncer (NCI), revisa este resumen de manera periódica y, en caso necesario, lo actualiza. Este resumen es el resultado de una revisión bibliográfica independiente y no constituye una declaración de política del NCI ni de los Institutos Nacionales de la Salud (NIH).
Cada mes, los integrantes de este consejo revisan los artículos publicados recientemente para determinar lo siguiente:
- Si el artículo se debe analizar en una reunión del consejo.
- Si conviene añadir texto acerca del artículo.
- Si se debe reemplazar o actualizar un artículo que ya se citó.
Los cambios en los resúmenes se deciden mediante consenso de los integrantes del consejo después de evaluar la solidez de la evidencia de los artículos publicados y determinar la forma de incorporar el artículo en el resumen.
Los revisores principales del sumario sobre Tratamiento del sarcoma de Ewing y los sarcomas indiferenciados de células redondas pequeñas de hueso y tejido blando son:
- Holcombe Edwin Grier, MD
- Andrea A. Hayes-Dixon, MD, FACS, FAAP (Howard University)
- Karen J. Marcus, MD, FACR (Dana-Farber of Boston Children's Cancer Center and Blood Disorders Harvard Medical School)
- William H. Meyer, MD
- Paul A. Meyers, MD (Memorial Sloan-Kettering Cancer Center)
- Thomas A. Olson, MD (Aflac Cancer and Blood Disorders Center of Children's Healthcare of Atlanta - Egleston Campus)
- Nita Louise Seibel, MD (National Cancer Institute)
- Malcolm A. Smith, MD, PhD (National Cancer Institute)
Cualquier comentario o pregunta sobre el contenido de este resumen se debe enviar al Servicio de Información de Cáncer del Instituto Nacional del Cáncer. Por favor, no enviar preguntas o comentarios directamente a los integrantes del consejo, ya que no responderán consultas de manera individual.
Niveles de evidencia
Algunas de las referencias bibliográficas de este resumen se acompañan del nivel de evidencia. El propósito de esto es ayudar al lector a evaluar la solidez de la evidencia que respalda el uso de ciertas intervenciones o abordajes. El consejo editorial del PDQ sobre el tratamiento pediátrico emplea un sistema de jerarquización formal para asignar los niveles de evidencia científica.
Permisos para el uso de este resumen
PDQ (Physician Data Query) es una marca registrada. Se autoriza el uso del texto de los documentos del PDQ; sin embargo, no se podrá identificar como un resumen de información sobre cáncer del PDQ del NCI, salvo que el resumen se reproduzca en su totalidad y se actualice de manera periódica. Por otra parte, se permitirá que un autor escriba una oración como “En el resumen del PDQ del NCI de información sobre la prevención del cáncer de mama se describen, de manera concisa, los siguientes riesgos: [incluir fragmento del resumen]”.
Se sugiere citar la referencia bibliográfica de este resumen del PDQ de la siguiente forma:
PDQ® sobre el tratamiento pediátrico. PDQ Tratamiento del sarcoma de Ewing y los sarcomas indiferenciados de células redondas pequeñas de hueso y tejido blando. Bethesda, MD: National Cancer Institute. Actualización: <MM/DD/YYYY>. Disponible en: https://www.cancer.gov/espanol/tipos/hueso/pro/tratamiento-ewing-pdq. Fecha de acceso: <MM/DD/YYYY>.
Las imágenes en este resumen se reproducen con autorización del autor, el artista o la editorial para uso exclusivo en los resúmenes del PDQ. La utilización de las imágenes fuera del PDQ requiere la autorización del propietario, que el Instituto Nacional del Cáncer no puede otorgar. Para obtener más información sobre el uso de las ilustraciones de este resumen o de otras imágenes relacionadas con el cáncer, consultar Visuals Online, una colección de más de 2000 imágenes científicas.
Cláusula sobre el descargo de responsabilidad
Según la solidez de la evidencia, las opciones de tratamiento se clasifican como “estándar” o “en evaluación clínica”. Estas clasificaciones no se deben utilizar para justificar decisiones sobre reembolsos de seguros. Para obtener más información sobre la cobertura de seguros, consultar la página Manejo de la atención del cáncer en Cancer.gov/espanol.
Comuníquese con el Instituto Nacional del Cáncer
Para obtener más información sobre las opciones para comunicarse con el NCI, incluso la dirección de correo electrónico, el número telefónico o el chat, consultar la página del Servicio de Información de Cáncer del Instituto Nacional del Cáncer.